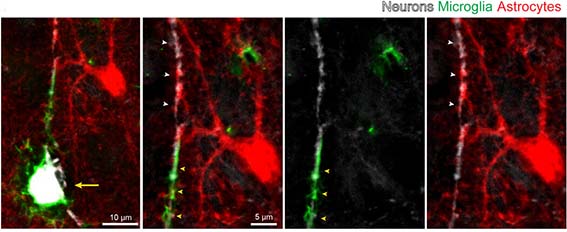
前回 [1] に引き続き、
先日、TMEM106Bに関する驚きの論文がCellに1報、Natureに2報掲載されました。 Cellの論文は、アメリカ・コロンビア大学のDr.Fitzpatric、カナダ・英コロンビア大学のDr.Mackenzieらの …
TMEM106Bに関する論文。
今回は、Nature誌に掲載された、イギリス・MRC研究所のDr. Goedert、Dr. Scheresらの研究グループからで、色々な神経変性疾患の他、健常高齢者の脳内でもTMEM106Bの凝集体が観察された、というcryo-EMでの解析 [2]。
Many age-dependent neurodegenerative diseases, like Alzheimer’s and Parkinson’s, are characterised by abundant inclusions of amyloid filaments. Filamentous inclusions of the proteins tau, amyloid-β (Aβ), α-synuclein and TDP-43 are the most common1,2. Here, we used electron cryo-microscopy (cryo-EM) structure determination to show that residues 120-254 of the lysosomal type II transmembrane protein 106B (TMEM106B) also form amyloid filaments in human brains. We determined the cryo-EM structures of TMEM106B filaments from a number of brain regions of 22 individuals with abundant amyloid deposits, including sporadic and inherited tauopathies, Aβ-amyloidoses, synucleinopathies and TDP-43 proteinopathies, as well as from the frontal cortex of 3 neurologically normal individuals with no or only few amyloid deposits. We observed three TMEM106B folds, with no clear relationships between folds and diseases. TMEM106B filaments correlated with the presence of a 29 kDa sarkosyl-insoluble fragment and globular cytoplasmic inclusions, as detected by an antibody specific for the C-terminal region of TMEM106B. The identification of TMEM106B filaments in the brains of older, but not younger, neurologically normal individuals indicates that they form in an age-dependent manner.
ざっくりおさらいですが、TMEM106Bは274aaのII型膜貫通型蛋白で、脳・心臓・甲状腺・副腎・精巣などに多く [3] 、脳内では神経・グリア・内皮細胞・ペリサイトなどのリソソームとエンドソームの膜に存在しています [4] 。
APPと同じように細胞膜の外側と膜内で2段階切断を受け、内宮側ドメインのC末側はリソソーム内に放出されます。
何らかのリソソームタンパク分解酵素として機能していると推測されていますが、どんな酵素なのかはまだはっきりとはわかっていません。
変性疾患との関係については私も何度かこのブログで取り上げましたが、
以前は全く別の疾患と考えられていたものが、共通のリスク因子や病理を持つことが多いという認識が広まり、神経変性疾患の枠組みも大きく変わってきていると感じます。 今回の論文は、frontotemporal lobar deg …
今回は、前頭側頭葉変性症(Frontotemporal lobar degeneration, FTLD)の約半数を占める 「TDP43凝集体を伴うFTLD(FTLD-TDP)」 …に深く関与する、 Progranuli …
今までは、FTD/ALS、LATE、Hippocampal sclerosisなど、TDP-43プロテイノパチーのリスク遺伝子として知られており、
同じくTDP-43プロテイノパチーのリスク遺伝子であるgranulin遺伝子(GRN)変異との関連が強く、これらの疾患のモジュレーターとして考えられていました。
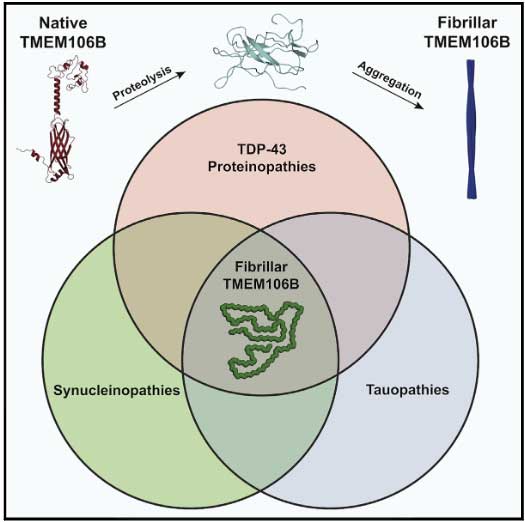
それが今回の3報では、TEME106B自身が人の脳内で凝集体を形成して沈着していることがわかり [1, 2, 5] 、
もしこれが他の蛋白のように変性疾患の病態に大きく関わっているようであれば、タウオパチー・シヌクレイノパチー・TDP-43プロテイノパチーに引き続き、「TMEM106Bプロテイノパチー」のような概念が出てくるかもしれません。
とゆーことで、今回は、Dr. Goedertらの論文のまとめ。

TMEM06B凝集体が、神経変性疾患および健常高齢者脳内で観察される
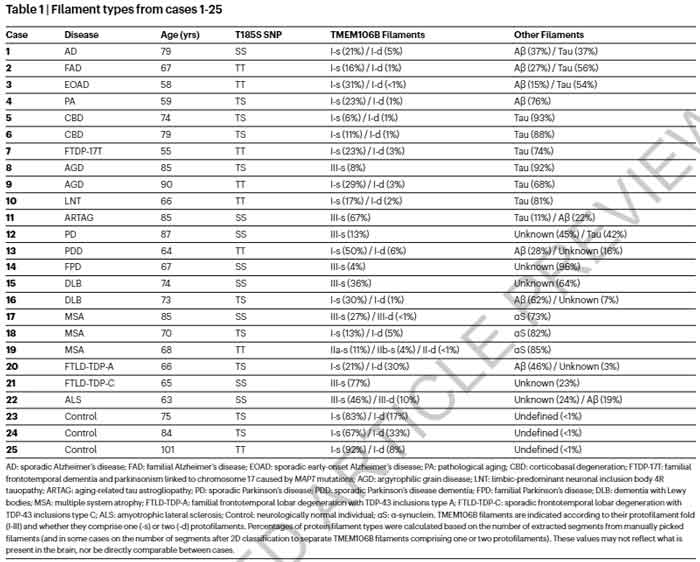
25症例でTMEM106B凝集体を検出
彼らは、25症例の脳から、α-Syn凝集体などで既に確立している方法でサルコシル不溶性蛋白凝集体を抽出しました。
TMEM106B凝集体は他の蛋白凝集体と同じようにfuzzy coat部分があり、コア部分は120-254aaで構成されていました。
これらのフィラメントは、
- アルツハイマー病(Alzheimer's disease, AD)
- 高齢病理脳(pathological aging, PA)
- 大脳皮質基底核変性症(corticobasal degeneration, CBD)
- 前頭側頭葉変性症(frontotemporal lobar degeneration, FTLD)
- TypeA
- TypeC
- FTDP-17T
- 嗜銀顆粒性認知症(argyrophilic Grain Dementia, AGD)
- limbic-predominant neuronal inclusion body 4R tauopathy (LNT)
- aging-related tau astrogliopathy (ARTAG)
等、色々な神経変性疾患の脳内の他、健常高齢者脳でも検出されました。
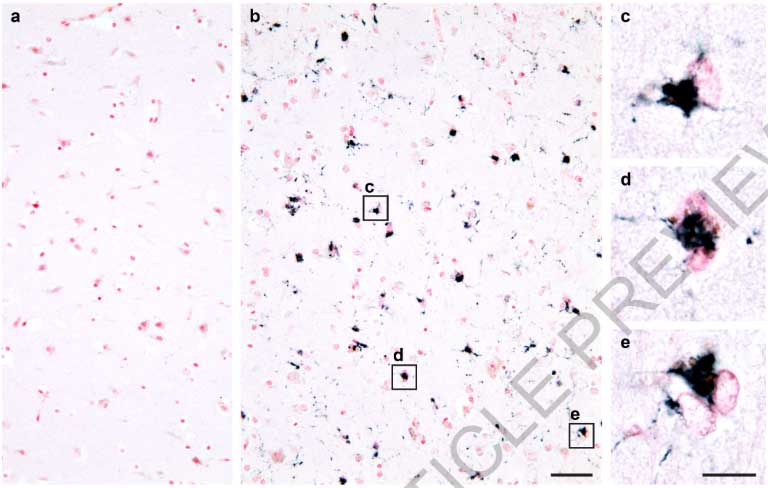
TMEM106B凝集体のcryo-EM
彼らは、抽出したTMEM106B凝集体をcryo-EMで解析しました。
Dr. Fitzpatrickらの報告と同じように、凝集体はTMEM106B(120-154aa)で構成されており、
3種類の異なるタイプのプロトフィラメントがありました。
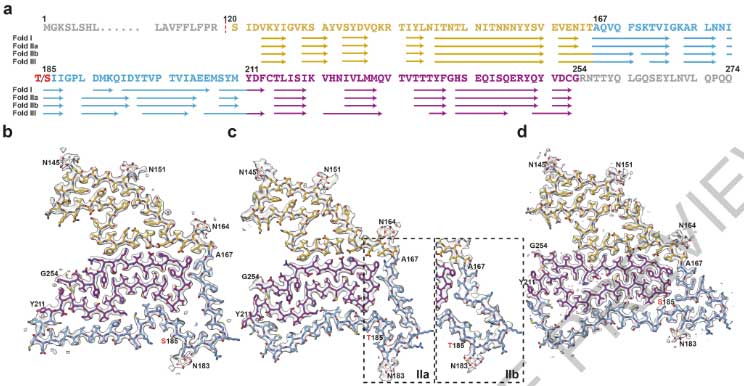
TMEM106Bプロトフィラメントは5層の折りたたみ構造になっていて、17のβ-strands(Dr. Fitzpatrickらは19と報告)がありました。
全ての層で、可能な側鎖は全てグリコシル化されていて、145, 151, 164, 183の糖鎖を反映すように大きな高密度領域がありました。
5番目のグリコシル化部位であるN256は、コアの外側にあり、C末側の20残基はおそらく崩れた構造をしていると予測されました。
もう少し詳しくみると、
- N末側のS120-T166は前2層の折りたたみコアを構成
- C末側のY211-G254は真ん中2層の折りたたみコアを構成
- 中間部のS166-M210は最後の折りたたみコアを構成し、4番目のグリコシル化部位(N183)を含む
という感じでした。
AlfaFoldでネイティブ構造を予測
今までTMEM106Bのネイティブ構造が明らかでなかったので、著者らはAlfaFoldでネイティブ構造を予測してみました [6] 。
アミロイドフィブリルの多くは天然変性(natively unfolded proteins)もしくは低複雑性ドメイン(low-complexity protein domains)関連のようだったけれども、
フィブリルコア部分のS120-G254シークエンスは免疫グロブリン様ドメイン(globular domain of the immunoglobulin-like β-sandwich fold)を有していました。
グリコシル化部位のN145, N151, N164, N183は折りたたみ構造の外側に位置していて、C214とC253の間でジスルフィド結合が起こると予測されました。
β-sandwichドメインはリンカー配列(flexible linker sequence)なしで一型膜貫通ヘリックス部分とつながっていました。
さらに、このドメインの後ろには疎水性パッチ(hydrophobic surface patch)があり、膜に近いところに位置していると考えられました。
以上のような配列から、TMEM106Bフィラメント内のN末側のS120の切断部位はリソソームの分解を受けにくい構造になっている事が予測されました。
この内腔側ドメインの切断は非典型的経路で起こっているのかもしれません。
健常人でも高齢者の脳内でのTMEM106Bの凝集体を観察
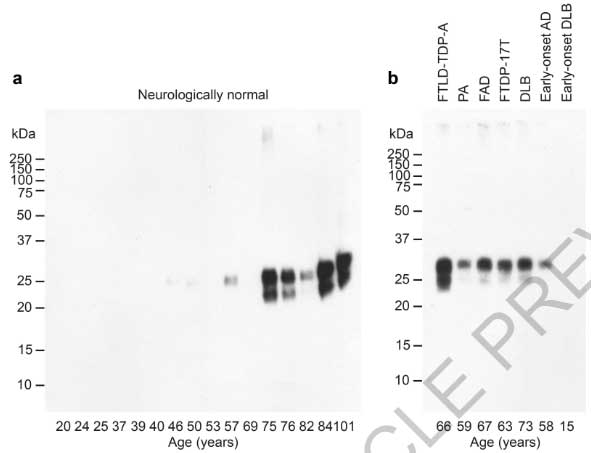
彼らは、20歳から101歳までの認知機能正常な人達の剖検脳からサルコシル不溶性画分を抽出し、TMEM1076Bの239-250aaを認識する抗体(TMEM239)でWBしてみました。
すると、29kDa 部位にバンドがみられ、17kDaのC末断端+12kDaのグリコシル化や他の翻訳後修飾修飾を受けたTMEM106Bと考えられました。
このバンドは46歳以下では検出されず、69歳以上の脳内で多く検出されました。
神経変性疾患の脳内でも、15歳で亡くなった早期発症レヴィ小体型認知症(dementia with Lewy bodies, DLB)の患者さん脳内では検出されず、
TMEM106B凝集体は年齢依存的に増加する可能性が示唆されました。
彼らは同じTMEM239抗体で免疫染色も行いましたが、WBの結果と同様に、高齢者脳内でTMEM106Bの凝集体が観察されました。

Many age-dependent neurodegenerative diseases, like Alzheimer’s and Parkinson’s, are characterised by abundant inclusions of amyloid filaments. Filamentous inclusions of the proteins tau, amyloid-β (Aβ), α-synuclein and TDP-43 are the most common1,2. Here, we used electron cryo-microscopy (cryo-EM) structure determination to show that residues 120-254 of the lysosomal type II transmembrane protein 106B (TMEM106B) also form amyloid filaments in human brains. We determined the cryo-EM structures of TMEM106B filaments from a number of brain regions of 22 individuals with abundant amyloid deposits, including sporadic and inherited tauopathies, Aβ-amyloidoses, synucleinopathies and TDP-43 proteinopathies, as well as from the frontal cortex of 3 neurologically normal individuals with no or only few amyloid deposits. We observed three TMEM106B folds, with no clear relationships between folds and diseases. TMEM106B filaments correlated with the presence of a 29 kDa sarkosyl-insoluble fragment and globular cytoplasmic inclusions, as detected by an antibody specific for the C-terminal region of TMEM106B. The identification of TMEM106B filaments in the brains of older, but not younger, neurologically normal individuals indicates that they form in an age-dependent manner.
My View
今回の論文ではどうやって凝集体がTEME106Bと判明したかは書いていなかったのですが、
前回紹介したCell論文 [1] のラストオーサーのDr.Fitzpatricはここのラボ出身なので、おそらく論文投稿前の段階から連絡をとりあって情報を共有していたんじゃないかと思います。
2002年にノーベル賞を受賞した島津製作所の田中氏が開発した方法でMSでの蛋白質解析が可能になったわけですが、こんな感じで重要蛋白を次々と発見していて、やっぱりすごい開発だったんだなーと思います。
最近元気がない日本ですが、先人達がコツコツ積み上げてきた素晴らしい発見・発明を足がかりにして、みんなで頑張っていきたいところです。
……と感慨に耽りながらも、今回の論文。
色々な変性疾患だけでなく、健常高齢者でもTMEM106B凝集体が観察されており、limbic-predominant age-related TDP-43 encephalopathy(LATE)や加齢関連タウアストログリオパチー(aging-related tau astrogliopathy, ARTAG)などと同じく加齢と密接な関係がありそうです。
免染の画像を見ると、この凝集体はタウ・α-Syn・TDP-43の凝集体とは全然違う形態をしているので、「TMEM106Bはそれのみで、他の蛋白とは異なる凝集体を形成するんだろう」という事が想像できます。
問題は「この凝集体が悪さをしているのかどうか」という事ですが、その機能についてはこれから続々と報告が上がってくるはずです。
剖検脳が手に入る人達は、既にTMEM106B抗体で染色したり、サルコシル不溶画分を抽出して解析したり細胞にかけたり脳に打ち込んだりし始めている事でしょう。
そこに自分が参加できないのは残念ですが(今やっている事で手一杯なので)、タイミングがあえば是非取り組んでみたいテーマだと思います。
References
- Chang A, Xiang X, Wang J, Lee C, Arakhamia T, Simjanoska M, Wang C, Carlomagno Y, Zhang G, Dhingra S, Thierry M, Perneel J, Heeman B, Forgrave LM, DeTure M, DeMarco ML, Cook CN, Rademakers R, Dickson DW, Petrucelli L, Stowell MHB, Mackenzie IRA, Fitzpatrick AWP. Homotypic fibrillization of TMEM106B across diverse neurodegenerative diseases. Cell. 2022 Mar 1:S0092-8674(22)00259-8. doi: 10.1016/j.cell.2022.02.026. Epub ahead of print. PMID: 35247328.
- Schweighauser M, Arseni D, Bacioglu M, Huang M, Lövestam S, Shi Y, Yang Y, Zhang W, Kotecha A, Garringer HJ, Vidal R, Hallinan GI, Newell KL, Tarutani A, Murayama S, Miyazaki M, Saito Y, Yoshida M, Hasegawa K, Lashley T, Revesz T, Kovacs GG, van Swieten J, Takao M, Hasegawa M, Ghetti B, Spillantini MG, Ryskeldi-Falcon B, Murzin AG, Goedert M, Scheres SHW. Age-dependent formation of TMEM106B amyloid filaments in human brains. Nature. 2022 Mar 28. doi: 10.1038/s41586-022-04650-z. Epub ahead of print. PMID: 35344985.
- http://www.proteinatlas.org/
- Nicholson AM, Rademakers R. What we know about TMEM106B in neurodegeneration. Acta Neuropathol. 2016 Nov;132(5):639-651. doi: 10.1007/s00401-016-1610-9. Epub 2016 Aug 20. PMID: 27543298; PMCID: PMC5074873.
- Jiang YX, Cao Q, Sawaya MR, Abskharon R, Ge P, DeTure M, Dickson DW, Fu JY, Ogorzalek Loo RR, Loo JA, Eisenberg DS. Amyloid fibrils in disease FTLD-TDP are composed of TMEM106B not TDP-43. Nature. 2022 Mar 28. doi: 10.1038/s41586-022-04670-9. Epub ahead of print. PMID: 35344984.
- Tunyasuvunakool K, Adler J, Wu Z, Green T, Zielinski M, Žídek A, Bridgland A, Cowie A, Meyer C, Laydon A, Velankar S, Kleywegt GJ, Bateman A, Evans R, Pritzel A, Figurnov M, Ronneberger O, Bates R, Kohl SAA, Potapenko A, Ballard AJ, Romera-Paredes B, Nikolov S, Jain R, Clancy E, Reiman D, Petersen S, Senior AW, Kavukcuoglu K, Birney E, Kohli P, Jumper J, Hassabis D. Highly accurate protein structure prediction for the human proteome. Nature. 2021 Aug;596(7873):590-596. doi: 10.1038/s41586-021-03828-1. Epub 2021 Jul 22. PMID: 34293799; PMCID: PMC8387240.