脳内での細胞達の動向をみるために、シングルセルRNAシークエンス(single-cell RNA-Seq, scRNA-Seq)が注目され、
これまで、マウス脳内やヒト脳内での各細胞のheterogeneityについて多数報告されてきました [1, 2, 3] 。
特にミクログリアでは、今までマウスとヒトとでだいぶプロファイルが違うと言われてきており [4]、
ミクログリアを扱う同僚の一人は、
「マウスでミクログリアの研究をしててもダメだ。」
と話していました。
シングルセルNRAシークエンスの単離法には、
細胞を単離する方法の他、
保存サンプルからの細胞核を単離して解析する方法(single-nucleus RNA-seq, snRNA-Seq)があり、
ヒト脳の実験の場合はだいたい凍結した剖検脳を解析するので、
剖検脳のシングルセルRNA-Seqには、主にこのsnRNA-seqが用いられていました。
今回、ベルギーのFlandersバイオテクノロジー研究所のDr. Strooper, Dr. Fiersらの研究グループは、
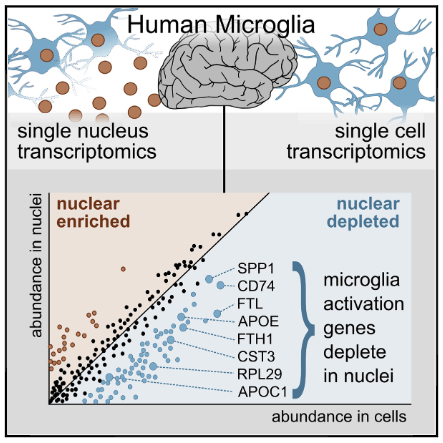
同じサンプルでscRNA-SeqとsnRNA-Seqの両方を行い、
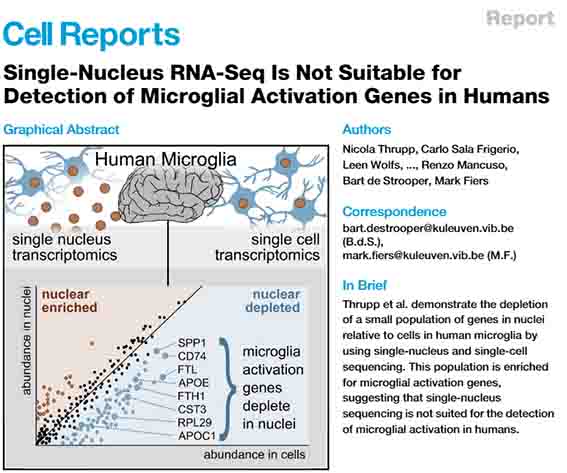
これらの異なった抽出法では、ミクログリアのプロファイルがだいぶ異なる事を報告しました [5]。
Single-Nucleus RNAシークエンスの落とし穴
著者らは、ヒトのてんかん患者さんの側頭葉のバイオプシーサンプル(n-4)を用いて、
scRNA-SeqとsnRNA-Seqの両方を行い、ミクログリアのプロファイルを比較した。
すると、scRNA-SeqとsnRNA-Seqでは、RNAプロファイルが色々異なっており、
snRNA-Seqではある特定の遺伝子発現が濃縮されていたり、別の遺伝子発現を拾えていなかったりした。
ADマウスモデルの活性型ミクログリアで上がっていると言われる遺伝子群を調べると、
- Disease-associated microglia (DAM) 関連遺伝子257種類中45遺伝子
- Activation response microglia (ARM) 関連遺伝子200種類中28遺伝子
が、snRNA-seqでは拾えておらず、
マウス脳内のミクログリア活性化遺伝子は、ヒトのミクログリア核内に少ないということがわかった。
eg.
- APOE
- CST3
- FTL
- SPP1
- B2M
- PLD3
- CD74
- FTH1
- RPL29
- APOC1
以前、Eggenらの研究グループが、
「scRNA-SeqとsnRAN-Seqのプロファイルがだいたい同じ」
という報告をしているので [6]、
著者らは彼らの生データを入手し、同様に解析した。
結果、フレッシュな核プロファイルと細胞プロファイルを比較すると、ほとんど差はなかった。
しかしながら、凍結した核とフレッシュな細胞のプロファイルを比較すると、それぞれのプロファイルに乖離があった。
My View
私自身はシングルセルRNA-Seqをしたことはないですが、
AD脳のシングルセルRAN-Seqの講演で、
DAMやらARMやら聞き慣れない単語が出てきていたので、
「一回ちゃんと勉強しないとなー」
と思っていました。
ヒト剖検脳を使う場合は、大抵凍結保存なので、今回の研究結果のインパクトは大きいと思います。
snRNA-SeqとscRNA-SeqでいくつかのRNAプロファイルが違うのは、最初RNA局在によるものかと思っていましたが、
今回の論文や既報では、
両方ともフレッシュサンプルを使うとsnRAN-SeqとscRNA-Seqでほとんど差がないとのこと。
…ということはRNA局在が関係しているわけではなくて、
死後変化や凍結による破壊等が関係しているということかな?
でも、AD等の神経変性疾患では、てんかん患者さんや脳腫瘍の患者さんみたいに脳バイオプシーをしたりする機会はないので、
この論文の内容が本当なら、
「神経変性疾患の脳内の状況を知るのに、シングルセルRNA-Seqによるアプローチはちょっと難しい」ということになります。
代替案としては、In situ spatial transcriptomics [7] とか、NanoString [8] とかになるのかな?
…という感じで、
凍結サンプルでのnsRNA-SeqとscRNA-Seqの結果に違いがでる原因はよくわかりませんでしたが、
とにかく、今までの報告をもう一度見直す必要がありそうだということと、
ヒトAD脳内のミクログリア等のプロファイルは今後も追求の余地がありそう、
という印象を持ちました。
References
- Mathys H, Davila-Velderrain J, Peng Z, Gao F, Mohammadi S, Young JZ, Menon M, He L, Abdurrob F, Jiang X, Martorell AJ, Ransohoff RM, Hafler BP, Bennett DA, Kellis M, Tsai LH. Single-cell transcriptomic analysis of Alzheimer's disease. Nature. 2019 Jun;570(7761):332-337. doi: 10.1038/s41586-019-1195-2. Epub 2019 May 1. Erratum in: Nature. 2019 Jun 17;: PMID:31042697; PMCID: PMC6865822.
- Del-Aguila JL, Li Z, Dube U, Mihindukulasuriya KA, Budde JP, Fernandez MV, Ibanez L, Bradley J, Wang F, Bergmann K, Davenport R, Morris JC, Holtzman DM, Perrin RJ, Benitez BA, Dougherty J, Cruchaga C, Harari O. A single-nuclei RNA sequencing study of Mendelian and sporadic AD in the human brain. Alzheimers Res Ther. 2019 Aug 9;11(1):71. doi: 10.1186/s13195-019-0524-x. PMID:31399126; PMCID: PMC6689177.
- Grubman A, Chew G, Ouyang JF, Sun G, Choo XY, McLean C, Simmons RK, Buckberry S, Vargas-Landin DB, Poppe D, Pflueger J, Lister R, Rackham OJL, Petretto E, Polo JM. A single-cell atlas of entorhinal cortex from individuals with Alzheimer's disease reveals cell-type-specific gene expression regulation. Nat Neurosci. 2019 Dec;22(12):2087-2097. doi: 10.1038/s41593-019-0539-4. PMID:31768052.
- Zhou Y, Song WM, Andhey PS, Swain A, Levy T, Miller KR, Poliani PL, Cominelli M, Grover S, Gilfillan S, Cella M, Ulland TK, Zaitsev K, Miyashita A, Ikeuchi T, Sainouchi M, Kakita A, Bennett DA, Schneider JA, Nichols MR, Beausoleil SA, Ulrich JD, Holtzman DM, Artyomov MN, Colonna M. Human and mouse single-nucleus transcriptomics reveal TREM2-dependent and TREM2-independent cellular responses in Alzheimer's disease. Nat Med. 2020 Jan;26(1):131-142. doi: 10.1038/s41591-019-0695-9. Epub 2020 Jan 13. Erratum in: Nat Med. 2020 Jun;26(6):981. PMID:31932797; PMCID: PMC6980793.
- Thrupp N, Sala Frigerio C, Wolfs L, Skene NG, Fattorelli N, Poovathingal S, Fourne Y, Matthews PM, Theys T, Mancuso R, de Strooper B, Fiers M. Single-Nucleus RNA-Seq Is Not Suitable for Detection of Microglial Activation Genes in Humans. Cell Rep. 2020 Sep 29;32(13):108189. doi: 10.1016/j.celrep.2020.108189. PMID:32997994; PMCID: PMC7527779.
- Gerrits E, Heng Y, Boddeke EWGM, Eggen BJL. Transcriptional profiling of microglia; current state of the art and future perspectives. Glia. 2020 Apr;68(4):740-755. doi: 10.1002/glia.23767. Epub 2019 Dec 17. PMID:31846124; PMCID: PMC7064956.
- Visualization and analysis of gene expression in tissue sections by spatial transcriptomics BY PATRIK L. STÅHL, FREDRIK SALMÉN, SANJA VICKOVIC, ANNA LUNDMARK, JOSÉ FERNÁNDEZ NAVARRO, JENS MAGNUSSON, STEFANIA GIACOMELLO, MICHAELA ASP, JAKUB O. WESTHOLM, MIKAEL HUSS, ANNELIE MOLLBRINK, STEN LINNARSSON, SIMONE CODELUPPI, ÅKE BORG, FREDRIK PONTÉN, PAUL IGOR COSTEA, PELIN SAHLÉN, JAN MULDER, OLAF BERGMANN, JOAKIM LUNDEBERG, JONAS FRISÉN SCIENCE01 JUL 2016 : 78-82
- Prokop S, Miller KR, Labra SR, Pitkin RM, Hoxha K, Narasimhan S, Changolkar L, Rosenbloom A, Lee VM, Trojanowski JQ. Impact of TREM2 risk variants on brain region-specific immune activation and plaque microenvironment in Alzheimer's disease patient brain samples. Acta Neuropathol. 2019 Oct;138(4):613-630. doi: 10.1007/s00401-019-02048-2. Epub 2019 Jul 26. PMID:31350575; PMCID: PMC6939638.