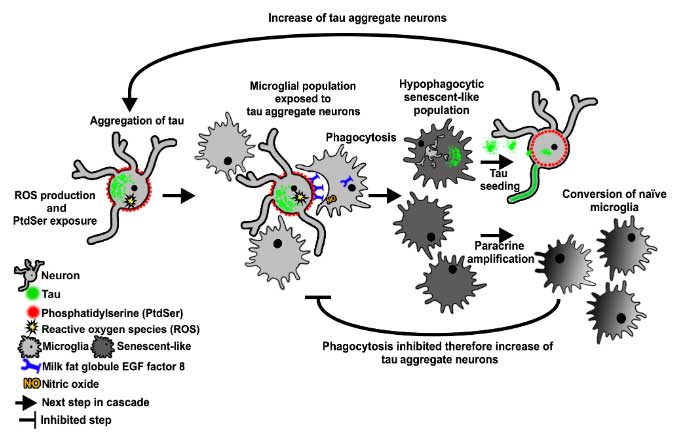
先日、TDP-43プロテイノパチーにおける cryptic exons の発現が筋萎縮性側索硬化症(Amyotrophic lateral sclerosis, ALS)などで発症前から増加するという論文 [1] を紹介しましたが、
TAR DNA-binding protein 43(TDP-43)には様々な機能が報告されていますが、その中でも重要なものとして、「遺伝子を正確に転写させる機能」があります。 筋萎縮性側索硬化症(Amyotrophic …
cryptic exonsは、ALSや前頭側頭型認知症(fronto-temporal dementia, FTD)だけではなく、アルツハイマー病 (Alzheimer's disease, AD) に合併した辺縁系優位型加齢性TDP-43脳症(limbic-predominant age-related TDP-43 encephalopathy, LATE)でも見られるようです。
今回は、ADに合併したLATE(LATE-AD)で、cryptic splicingを観察した論文2報を紹介。
cryptic splicingは、アルツハイマー病に合併したLATEでも増えている
Nuclear clearance and cytoplasmic accumulations of the RNA-binding protein TDP-43 are pathological hallmarks in almost all patients with amyotrophic lateral sclerosis (ALS) and up to 50% of patients with frontotemporal dementia (FTD) and Alzheimer’s disease. In Alzheimer’s disease, TDP-43 pathology is predominantly observed in the limbic system and correlates with cognitive decline and reduced hippocampal volume. Disruption of nuclear TDP-43 function leads to abnormal RNA splicing and incorporation of erroneous cryptic exons in numerous transcripts including Stathmin-2 (STMN2, also known as SCG10) and UNC13A, recently reported in tissues from patients with ALS and FTD. Here, we identify both STMN2 and UNC13A cryptic exons in Alzheimer’s disease patients, that correlate with TDP-43 pathology burden, but not with amyloid-β or tau deposits. We also demonstrate that processing of the STMN2 pre-mRNA is more sensitive to TDP-43 loss of function than UNC13A. In addition, full-length RNAs encoding STMN2 and UNC13A are suppressed in large RNA-seq datasets generated from Alzheimer’s disease post-mortem brain tissue. Collectively, these results open exciting new avenues to use STMN2 and UNC13A as potential therapeutic targets in a broad range of neurodegenerative conditions with TDP-43 proteinopathy including Alzheimer’s disease.
LATE-NC, the neuropathologic changes of limbic-predominant age-related TAR DNA-binding protein 43 kDa (TDP-43) encephalopathy are frequently associated with Alzheimer’s disease (AD) and cognitive impairment in older adults. The association of TDP-43 proteinopathy with AD neuropathologic changes (ADNC) and its impact on specific cognitive domains are not fully understood and whether loss of TDP-43 function occurs early in the aging brain remains unknown. Here, using a large set of autopsies from the Baltimore Longitudinal Study of Aging (BLSA) and another younger cohort, we were able to study brains from subjects 21–109 years of age. Examination of these brains show that loss of TDP-43 splicing repression, as judged by TDP-43 nuclear clearance and expression of a cryptic exon in HDGFL2, first occurs during the 6th decade, preceding by a decade the appearance of TDP-43+ neuronal cytoplasmic inclusions (NCIs). We corroborated this observation using a monoclonal antibody to demonstrate a cryptic exon-encoded neoepitope within HDGFL2 in neurons exhibiting nuclear clearance of TDP-43. TDP-43 nuclear clearance is associated with increased burden of tau pathology. Age at death, female sex, high CERAD neuritic plaque score, and high Braak neurofibrillary stage significantly increase the odds of LATE-NC. Faster rates of cognitive decline on verbal memory (California Verbal Learning Test immediate recall), visuospatial ability (Card Rotations Test), mental status (MMSE) and semantic fluency (Category Fluency Test) were associated with LATE-NC. Notably, the effects of LATE-NC on verbal memory and visuospatial ability are independent of ADNC. However, the effects of TDP-43 nuclear clearance in absence of NCI on the longitudinal trajectories and levels of cognitive measures are not significant. These results establish that loss of TDP-43 splicing repression is an early event occurring in the aging population during the development of TDP-43 proteinopathy and is associated with increased tau pathology. Furthermore, LATE-NC correlates with high levels of ADNC but also has an impact on specific memory and visuospatial functions in aging that is independent of AD.
stathmin‑2とUNC13Aのcryptic splicingのmRNAsが、LATE-ADで増えている。
1報目の論文は、アメリカ・ハーバード大学のDr. Das、Lagier-Tourenne、メイヨー・クリニックのDr. Ertekin-Tanerらのグループから。
「TDP-43病理があるAD症例で、STMN2やUNC13Aのcryptic exonsの発現が増えている」
という事を報告しました [2]

Nuclear clearance and cytoplasmic accumulations of the RNA-binding protein TDP-43 are pathological hallmarks in almost all patients with amyotrophic lateral sclerosis (ALS) and up to 50% of patients with frontotemporal dementia (FTD) and Alzheimer’s disease. In Alzheimer’s disease, TDP-43 pathology is predominantly observed in the limbic system and correlates with cognitive decline and reduced hippocampal volume. Disruption of nuclear TDP-43 function leads to abnormal RNA splicing and incorporation of erroneous cryptic exons in numerous transcripts including Stathmin-2 (STMN2, also known as SCG10) and UNC13A, recently reported in tissues from patients with ALS and FTD. Here, we identify both STMN2 and UNC13A cryptic exons in Alzheimer’s disease patients, that correlate with TDP-43 pathology burden, but not with amyloid-β or tau deposits. We also demonstrate that processing of the STMN2 pre-mRNA is more sensitive to TDP-43 loss of function than UNC13A. In addition, full-length RNAs encoding STMN2 and UNC13A are suppressed in large RNA-seq datasets generated from Alzheimer’s disease post-mortem brain tissue. Collectively, these results open exciting new avenues to use STMN2 and UNC13A as potential therapeutic targets in a broad range of neurodegenerative conditions with TDP-43 proteinopathy including Alzheimer’s disease.
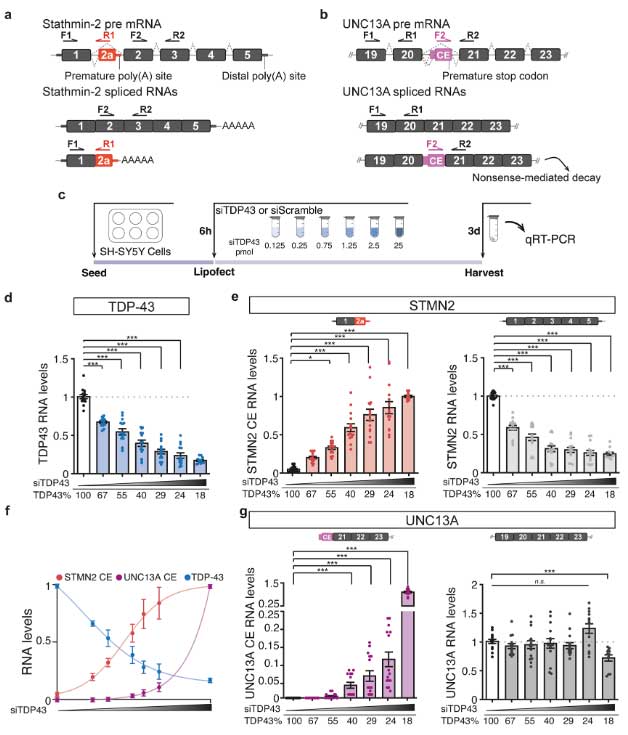
TDP-43を欠失すると、Stathmin-2とUNC13Aのcryptic splicingが増える
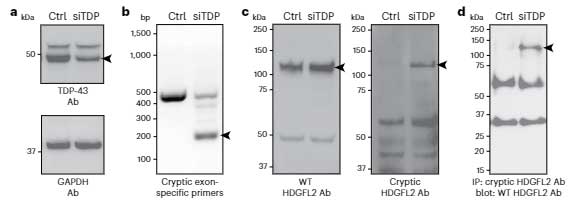
彼らはまず、TDP-43の欠失で既報どおりにStathmin-2とcryptic splicingが増えるのかどうか確認しました。
SH-SY5Y細胞のTDP-43をsiRNAでノックダウンすると、TDP-43の量に反比例して、STMN2とUNC13Aのcryptic splicingが増えていました。
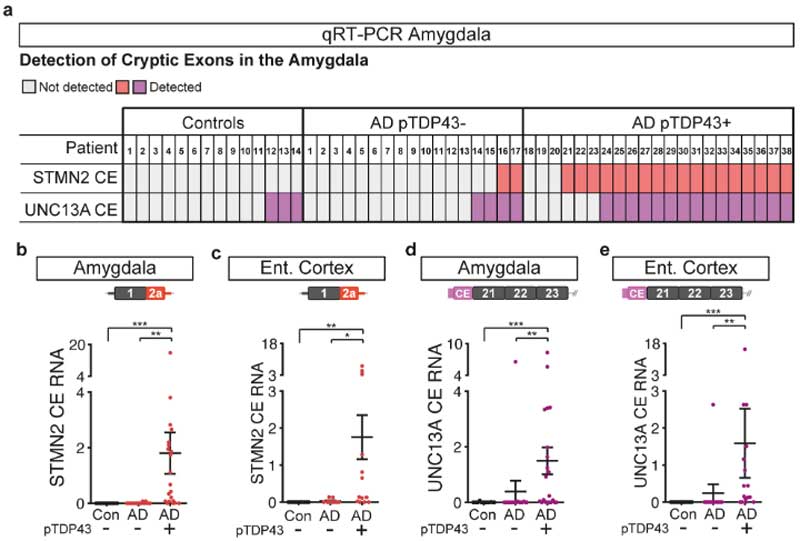
LATE-ADではSTMN2とUNC13Aのcryptic exonsの転写が増えている
彼らは次に剖検脳で、LATE病理を多く認める扁桃体や嗅内皮質でのSTMN2とUNC13Aのcryptic exonsの転写を調べました。
すると、健常人(Controls)やAD病理のみ(AD pTDP43-)の症例と比べて、LATE-AD(AD pTDP-43+)の症例で、STMN2とUNC13Aのcryptic exonsの転写が増えていました。
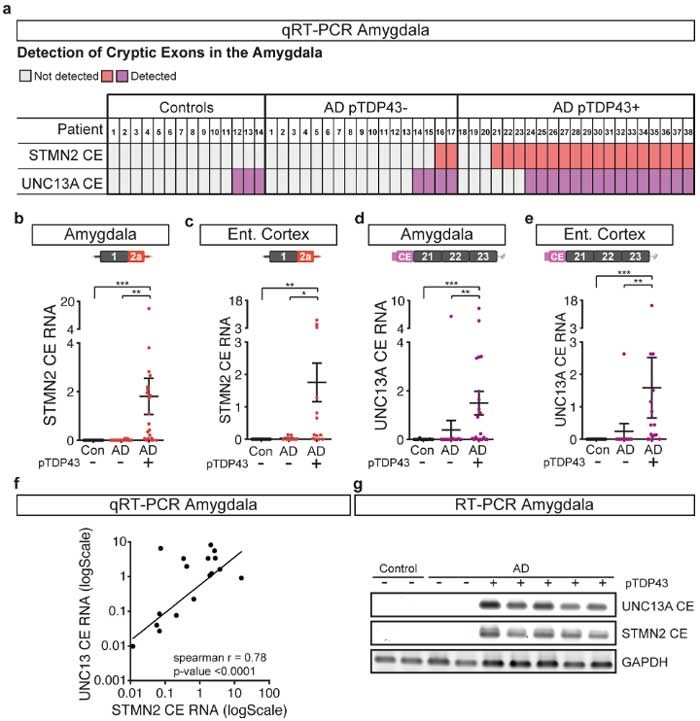
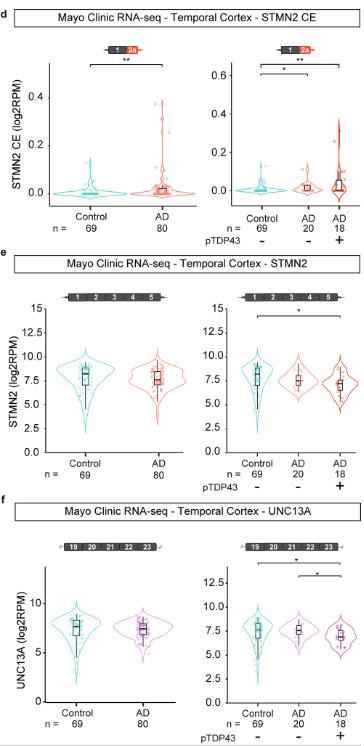
RNA-seqで調べると、STMN2とUNC13Aの全長をコードするRNAは減少しており、正常なタンパクの産生が落ちて機能が喪失している可能性が示唆されました。
stathmin-2とUNC13Aのcryptic splicingは、AD-LATEで増えている
2報目の論文は、アメリカ・ジョーンズホプキンス大学のDr. Troncosoらのグループから。
「ADの一部の症例では、LATE病理が生じる前から核内TDP-43の喪失が起こっており、その部位ではHDGFL2のcryptic exonsの発現が増えている」
という事を報告しました [3]

Nuclear clearance and cytoplasmic accumulations of the RNA-binding protein TDP-43 are pathological hallmarks in almost all patients with amyotrophic lateral sclerosis (ALS) and up to 50% of patients with frontotemporal dementia (FTD) and Alzheimer’s disease. In Alzheimer’s disease, TDP-43 pathology is predominantly observed in the limbic system and correlates with cognitive decline and reduced hippocampal volume. Disruption of nuclear TDP-43 function leads to abnormal RNA splicing and incorporation of erroneous cryptic exons in numerous transcripts including Stathmin-2 (STMN2, also known as SCG10) and UNC13A, recently reported in tissues from patients with ALS and FTD. Here, we identify both STMN2 and UNC13A cryptic exons in Alzheimer’s disease patients, that correlate with TDP-43 pathology burden, but not with amyloid-β or tau deposits. We also demonstrate that processing of the STMN2 pre-mRNA is more sensitive to TDP-43 loss of function than UNC13A. In addition, full-length RNAs encoding STMN2 and UNC13A are suppressed in large RNA-seq datasets generated from Alzheimer’s disease post-mortem brain tissue. Collectively, these results open exciting new avenues to use STMN2 and UNC13A as potential therapeutic targets in a broad range of neurodegenerative conditions with TDP-43 proteinopathy including Alzheimer’s disease.
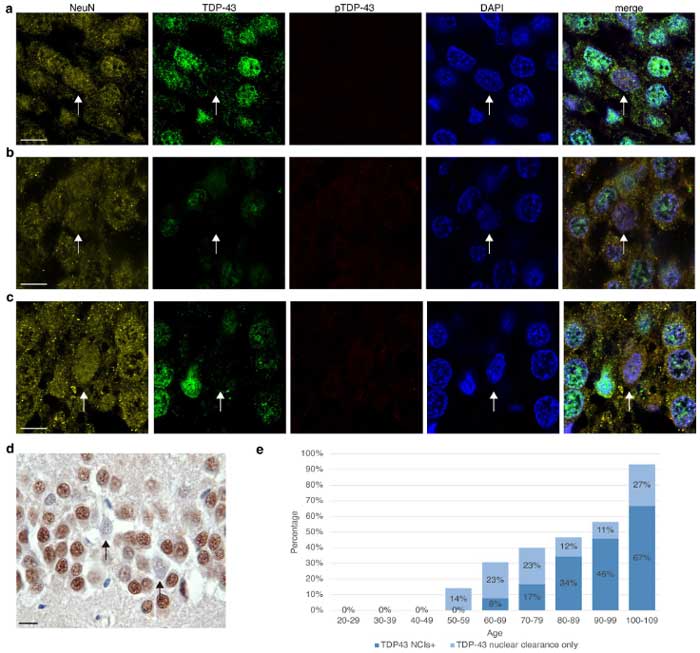
彼らは、Baltimore Longitudinal Study of Aging(BLSA)等のコホートに登録されている剖検脳を調べました。
TDP-43抗体で染色すると、TDP-43の神経細胞質内封入体(neurocytoplasmic inclusions, NCIs)は60代から徐々に現れますが、その10年前の50代から核内のTDP-43喪失を認める症例が出現しており、TDP-43の機能低下が起こっている可能性が示唆されました。
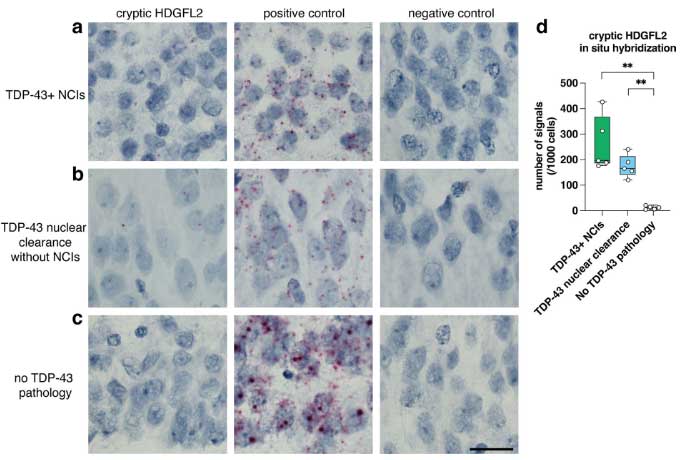
また、in situ hybridizationで確認したところ、TDP-43 NCIsがある症例や、NCIsはないけれども核内TDP-43の喪失が起こっている症例では、HDGFL2のcryptic splicingが増えていました。
My View
TDP-43は2006年にALS/FTLDの主要病理タンパクとして同定されました [4]。
統計的にはALSの95%、FTDの45%にTDP-43のNCIsを認め [5] 、現在はTDP-43プロテイノパチーという概念が提唱されています。
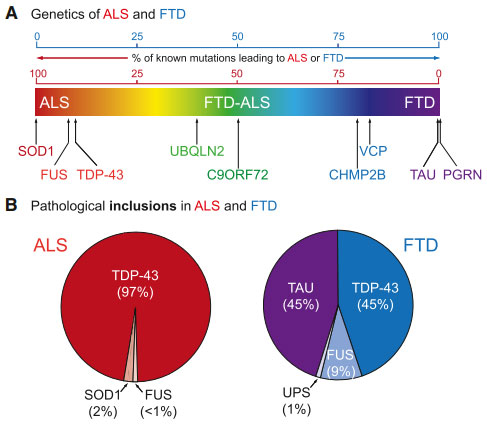
それからもうすぐ20年になろうとしていますが、その間、TDP-43の生理機構や、核内からTDP-43が喪失することによる機能障害の機構などが次々と解明されてきました。
一方、TDP-43病理は一般の高齢者脳内でも増える事がわかり、LATEと名付けられました [6]。
このLATE病理は、ALS/FTLDで認めるTDP-43 NCIsに酷似しており、病理出現のメカニズムも共通する部分が多そうに思います。
今回は、LATEでもTDP-43 NCIsを認める前から核内TDP-43の喪失が起こっていそうなこと、ALS/FTLDで認めるようなcryptic splicingがLATEでも起こっていそうな事などがわかりました。
去年も、TDP-43プロテイノパチーと関連の深い、TMEM106B凝集体がLATE症例にも多く認めるという論文が出ていたし [7] 、
TMEM106Bは、後期エンドソーム/リソソームの膜タンパクで、TDP-43プロテイノパチーのリスク多型となる遺伝子の一つでもあり [1]、以前から注目されていました。 特に昨年、FTLD-TDPや、普通のAging脳内 …
やっぱりALS/FTLDとLATEには共通のメカニズムが存在していそうです。。。
LATEの最も大きなリスクは「加齢」ですが、この「加齢とTDP-43の機能喪失を繋ぐメカニズム」はなんでしょうか?
それがわかったら凄くおもしろそうだなー、と思うのですが……それが解明される日もそう遠くはないかもしれません。
Glossary
TDP-43とcryptic exon
TDP-43には2つのRNA認識モチーフがあり、スプライシングを抑制することでRNA代謝を直接調節している。
この、TDP-43のスプライシング抑制機能が働かなくなると、間違ってイントロン配列の一部が転写される事があり、この部分の配列を cryptic exons と呼ぶ(日本語では儀エクソンや隠れエクソンなどと訳される。)[8]。
これらのイントロン部分は、本来は転写されない部分であるので、フレームシフトや、未成熟終止コドンや未成熟ポリアデニル化が含まれている事が多く、この部分が誤って転写されると、mRNA品質管理機構のナンセンスコドン介在的mRNA分解(nonsense-mediated decay, NMD)や、その他のRNA分解機構によって分解される [9, 10]。
これにより、本来翻訳されるべきタンパクの量が減り、機能障害をきたす可能性がある [11, 12, 13] 。
TDP-43の機能欠失により、cryptic exonが翻訳され、本来のタンパク量が減ってALSの病態をきたすタンパクとして、Stathmin-2 [14, 15] や UNC13A [16] がよく調べられている。
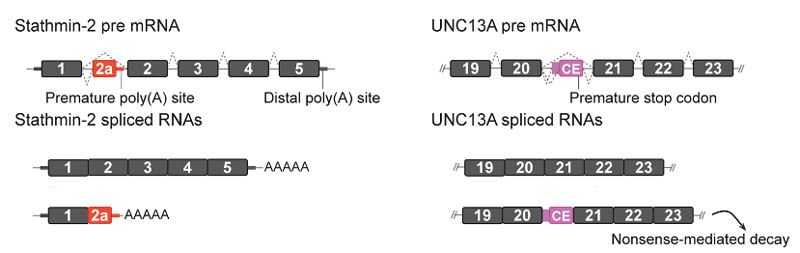
ナンセンスコドン介在的mRNA分解(nonsense-mediated decay, NMD) [17]
真核生物がもつnRNA品質管理機構の一つ。
ゲノムの突然変異や、転写・スプライシング異常などによって、翻訳領域に未成熟終止コドンが生じた場合、最初の翻訳段階で認識し、異常mRNAが翻訳される前に選択的に分解処理される。
この機構をナンセンスコドン介在的mRNA分解(nonsense-mediated decay, NMD)と呼ぶ。
NMDに関係するタンパクとして、酵母菌では、UPF1, UPF2, UPF3(ヒトではUPF3AとUPF3B)の3つが主要な因子と考えられている。
哺乳類では、UPF2とUPF3はエキソンジャンクション複合体の一部であり、スプライシング後にNMDにおいて機能を持つ他のタンパク質(eIF4AIII、MLN51、Y14/MAGOHヘテロダイマー)とともにmRNAと結合する。
UPF1のリン酸化反応はSMG-1、SMG-5、SMG-6、SMG-7といったタンパク質により制御されている。
References
- Irwin KE, Jasin P, Braunstein KE, Sinha IR, Garret MA, Bowden KD, Chang K, Troncoso JC, Moghekar A, Oh ES, Raitcheva D, Bartlett D, Miller T, Berry JD, Traynor BJ, Ling JP, Wong PC. A fluid biomarker reveals loss of TDP-43 splicing repression in presymptomatic ALS-FTD. Nat Med. 2024 Jan 26. doi: 10.1038/s41591-023-02788-5. Epub ahead of print. PMID: 38278991
- Agra Almeida Quadros AR, Li Z, Wang X, Ndayambaje IS, Aryal S, Ramesh N, Nolan M, Jayakumar R, Han Y, Stillman H, Aguilar C, Wheeler HJ, Connors T, Lopez-Erauskin J, Baughn MW, Melamed Z, Beccari MS, Olmedo Martínez L, Canori M, Lee CZ, Moran L, Draper I, Kopin AS, Oakley DH, Dickson DW, Cleveland DW, Hyman BT, Das S, Ertekin-Taner N, Lagier-Tourenne C. Cryptic splicing of stathmin-2 and UNC13A mRNAs is a pathological hallmark of TDP-43-associated Alzheimer's disease. Acta Neuropathol. 2024 Jan 4;147(1):9. doi: 10.1007/s00401-023-02655-0. PMID: 38175301; PMCID: PMC10766724.
- Chang K, Ling JP, Redding-Ochoa J, An Y, Li L, Dean SA, Blanchard TG, Pylyukh T, Barrett A, Irwin KE, Moghekar A, Resnick SM, Wong PC, Troncoso JC. Loss of TDP-43 splicing repression occurs early in the aging population and is associated with Alzheimer's disease neuropathologic changes and cognitive decline. Acta Neuropathol. 2023 Dec 22;147(1):4. doi: 10.1007/s00401-023-02653-2. PMID: 38133681.
- Neumann M, Sampathu DM, Kwong LK, Truax AC, Micsenyi MC, Chou TT, Bruce J, Schuck T, Grossman M, Clark CM, McCluskey LF, Miller BL, Masliah E, Mackenzie IR, Feldman H, Feiden W, Kretzschmar HA, Trojanowski JQ, Lee VM. Ubiquitinated TDP-43 in frontotemporal lobar degeneration and amyotrophic lateral sclerosis. Science. 2006 Oct 6;314(5796):130-3. doi: 10.1126/science.1134108. PMID: 17023659.
- Ling SC, Polymenidou M, Cleveland DW. Converging mechanisms in ALS and FTD: disrupted RNA and protein homeostasis. Neuron. 2013 Aug 7;79(3):416-38. doi: 10.1016/j.neuron.2013.07.033. PMID: 23931993; PMCID: PMC4411085.
- Nelson PT, Dickson DW, Trojanowski JQ, Jack CR, Boyle PA, Arfanakis K, Rademakers R, Alafuzoff I, Attems J, Brayne C, Coyle-Gilchrist ITS, Chui HC, Fardo DW, Flanagan ME, Halliday G, Hokkanen SRK, Hunter S, Jicha GA, Katsumata Y, Kawas CH, Keene CD, Kovacs GG, Kukull WA, Levey AI, Makkinejad N, Montine TJ, Murayama S, Murray ME, Nag S, Rissman RA, Seeley WW, Sperling RA, White CL 3rd, Yu L, Schneider JA. Limbic-predominant age-related TDP-43 encephalopathy (LATE): consensus working group report. Brain. 2019 Jun 1;142(6):1503-1527. doi: 10.1093/brain/awz099. Erratum in: Brain. 2019 Jul 1;142(7):e37. PMID: 31039256; PMCID: PMC6536849.
- Neumann M, Perneel J, Cheung S, Van den Broeck M, Nygaard H, Hsiung GR, Wynants S, Heeman B, Rademakers R, Mackenzie IRA. Limbic-predominant age-related TDP-43 proteinopathy (LATE-NC) is associated with abundant TMEM106B pathology. Acta Neuropathol. 2023 Jul;146(1):163-166. doi: 10.1007/s00401-023-02580-2. Epub 2023 May 12. PMID: 37171635.
- Ling JP, Pletnikova O, Troncoso JC, Wong PC. TDP-43 repression of nonconserved cryptic exons is compromised in ALS-FTD. Science. 2015 Aug 7;349(6248):650-5. doi: 10.1126/science.aab0983. PMID: 26250685; PMCID: PMC4825810.
- Humphrey J, Emmett W, Fratta P, Isaacs AM, Plagnol V. Quantitative analysis of cryptic splicing associated with TDP-43 depletion. BMC Med Genomics. 2017 May 26;10(1):38. doi: 10.1186/s12920-017-0274-1. PMID: 28549443; PMCID: PMC5446763.
- Mehta PR, Brown AL, Ward ME, Fratta P. The era of cryptic exons: implications for ALS-FTD. Mol Neurodegener. 2023 Mar 15;18(1):16. doi: 10.1186/s13024-023-00608-5. PMID: 36922834; PMCID: PMC10018954.
- Mehta PR, Brown AL, Ward ME, Fratta P. The era of cryptic exons: implications for ALS-FTD. Mol Neurodegener. 2023 Mar 15;18(1):16. doi: 10.1186/s13024-023-00608-5. PMID: 36922834; PMCID: PMC10018954.
- Seddighi S, Qi YA, Brown AL, Wilkins OG, Bereda C, Belair C, Zhang YJ, Prudencio M, Keuss MJ, Khandeshi A, Pickles S, Kargbo-Hill SE, Hawrot J, Ramos DM, Yuan H, Roberts J, Sacramento EK, Shah SI, Nalls MA, Colón-Mercado JM, Reyes JF, Ryan VH, Nelson MP, Cook CN, Li Z, Screven L, Kwan JY, Mehta PR, Zanovello M, Hallegger M, Shantaraman A, Ping L, Koike Y, Oskarsson B, Staff NP, Duong DM, Ahmed A, Secrier M, Ule J, Jacobson S, Reich DS, Rohrer JD, Malaspina A, Dickson DW, Glass JD, Ori A, Seyfried NT, Maragkakis M, Petrucelli L, Fratta P, Ward ME. Mis-spliced transcripts generate de novo proteins in TDP-43-related ALS/FTD. Sci Transl Med. 2024 Jan 26:eadg7162. doi: 10.1126/scitranslmed.adg7162. Epub ahead of print. PMID: 38277467.
- Cao MC, Ryan B, Wu J, Curtis MA, Faull RLM, Dragunow M, Scotter EL. A panel of TDP-43-regulated splicing events verifies loss of TDP-43 function in amyotrophic lateral sclerosis brain tissue. Neurobiol Dis. 2023 Sep;185:106245. doi: 10.1016/j.nbd.2023.106245. Epub 2023 Jul 30. PMID: 37527763.
- Klim JR, Williams LA, Limone F, Guerra San Juan I, Davis-Dusenbery BN, Mordes DA, Burberry A, Steinbaugh MJ, Gamage KK, Kirchner R, Moccia R, Cassel SH, Chen K, Wainger BJ, Woolf CJ, Eggan K. ALS-implicated protein TDP-43 sustains levels of STMN2, a mediator of motor neuron growth and repair. Nat Neurosci. 2019 Feb;22(2):167-179. doi: 10.1038/s41593-018-0300-4. Epub 2019 Jan 14. PMID: 30643292; PMCID: PMC7153761.
- Melamed Z, López-Erauskin J, Baughn MW, Zhang O, Drenner K, Sun Y, Freyermuth F, McMahon MA, Beccari MS, Artates JW, Ohkubo T, Rodriguez M, Lin N, Wu D, Bennett CF, Rigo F, Da Cruz S, Ravits J, Lagier-Tourenne C, Cleveland DW. Premature polyadenylation-mediated loss of stathmin-2 is a hallmark of TDP-43-dependent neurodegeneration. Nat Neurosci. 2019 Feb;22(2):180-190. doi: 10.1038/s41593-018-0293-z. Epub 2019 Jan 14. PMID: 30643298; PMCID: PMC6348009.
- Brown AL, Wilkins OG, Keuss MJ, Hill SE, Zanovello M, Lee WC, Bampton A, Lee FCY, Masino L, Qi YA, Bryce-Smith S, Gatt A, Hallegger M, Fagegaltier D, Phatnani H; NYGC ALS Consortium; Newcombe J, Gustavsson EK, Seddighi S, Reyes JF, Coon SL, Ramos D, Schiavo G, Fisher EMC, Raj T, Secrier M, Lashley T, Ule J, Buratti E, Humphrey J, Ward ME, Fratta P. TDP-43 loss and ALS-risk SNPs drive mis-splicing and depletion of UNC13A. Nature. 2022 Mar;603(7899):131-137. doi: 10.1038/s41586-022-04436-3. Epub 2022 Feb 23. PMID: 35197628; PMCID: PMC8891020.
- Lykke-Andersen S, Jensen TH. Nonsense-mediated mRNA decay: an intricate machinery that shapes transcriptomes. Nat Rev Mol Cell Biol. 2015 Nov;16(11):665-77. doi: 10.1038/nrm4063. Epub 2015 Sep 23. PMID: 26397022.