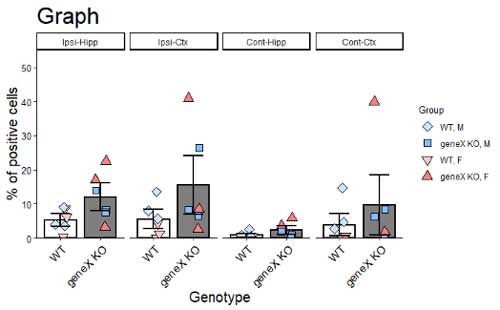
下記のようなグラフがあり、
これにプロットを重ねたい……でもただプロットするだけじゃなくて、各グループ内にいるオスとメスがプロットの形と色で区別できるようにしたい。
そんな場合の備忘録↓
グループ内でサブブループによってプロットの色と形を変える
1.サブグループの列を作り、レベル指定する
まずは、プロットの色形を変えるためのサブグループ毎のデータを指定する。
今回の例では、"Data" というデータフレーム内の "Group" という列に、
- WT, M
- GeneX KO, M
- WT, F
- GeneX KO, F
の4つのサブグループをつくり、factor型にしてレベルを指定しておく。
(ここでレベル指定しておくと、希望通りの順番に並んでくれる。)
Data$Group <- factor(Data$Group, levels = c("WT, M", "geneX KO, M", "WT, F", "geneX KO, F"))プロットの色と形を指定する
まずは ggplot でグラフを作り、
geom_point()
aes(shape = )そして
scale_shape_manual(values = c(形1, 形2, 形3, 形4))
scale_fill_manual(values = c(色1, 色2, 色3, 色4))今回は、上記4種類のサブグループなので指定する色形も4種類となる。
Data %>%
ggplot(
aes(x = Genotype,
y = FN,
fill = Group,
group = Genotype
)
) +
stat_summary(
fun = "mean", # 棒グラフの上底は平均値
geom = "bar", # グラフは棒グラフ
width = .8, # 棒グラフの幅は 0.8
colour = "black", # 色は黒
position = "dodge", # 棒グラフをジェノタイピング毎に横に並べる
size = 1, # 棒グラフの線の太さは1
fill = c("#fefefe", "#7f7f7f", "#fefefe", "#7f7f7f", "#fefefe", "#7f7f7f", "#fefefe", "#7f7f7f") # 棒グラフは白とグレーの繰り返し
) +
geom_point( # プロットの指定
position = position_jitterdodge( # プロットのポジションを少しずらして重なりを回避する
jitter.width = 1, # プロットの位置横に1ずらず
jitter.height = 0 # プロットの縦の位置はずらしたらダメ
),
size = 3.5, # プロットの大きさは3.5
colour = "black", # プロットの外線の色は黒
aes(shape = Group) # プロットは "Group" 毎に形を変える
) +
scale_shape_manual( # プロットの形を指定
values = c(23, 22, 25, 24)
) +
scale_fill_manual( # プロットの中の色を指定
values = c("#cce5ff", "#7fbfff", "#ffcccc", "#ff7f7f") #
)プロットの形は、下記参照。
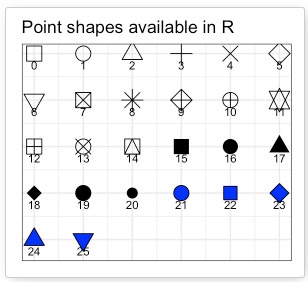
- 0-4 は白抜き。
- 5-20 は黒で塗りつぶし。
- 21-25 はマニュアルで色を設定。
私は棒グラフの色と合わせたかったので、23, 22, 25, 24 を選択。
プロットの外枠の色は
colour = "色の名称もしくは色番号"プロットの内部の色は
scale_fill_manual(values = c("色番号"))で指定される。
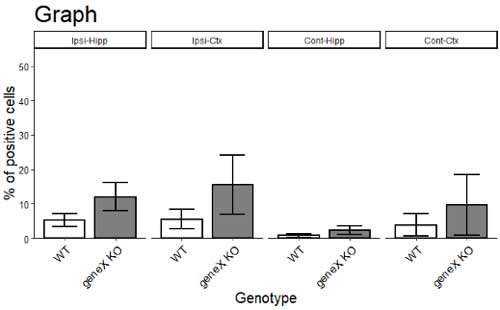
できあがり↓

・
・
・
全体のコードは下記 ↓
Data <- Data %>%
Data$Group <- factor(Data$Group, levels = c("WT, M", "geneX KO, M", "WT, F", "geneX KO, F"))
Data %>%
ggplot(
aes(x = Genotype,
y = FN,
fill = Group,
group = Genotype
)
) +
stat_summary(
fun = "mean",
geom = "bar",
width = .8,
colour = "black",
position = "dodge",
size = 1,
fill = c("#fefefe", "#7f7f7f", "#fefefe", "#7f7f7f", "#fefefe", "#7f7f7f", "#fefefe", "#7f7f7f")
) +
geom_point(
position = position_jitterdodge(
jitter.width = 1,
jitter.height = 0
),
size = 3.5,
colour = "black",
aes(shape = Group)
) +
scale_shape_manual(
values = c(23, 22, 25, 24)
) +
scale_fill_manual(
values = c("#cce5ff", "#7fbfff", "#ffcccc", "#ff7f7f")
) +
stat_summary(
fun = "mean",
fun.min = function(x)mean(x) - sd(x)/sqrt(3),
fun.max = function(x)mean(x) + sd(x)/sqrt(3),
geom = "errorbar", position = position_dodge(.8),
width = .5, size = 1,
colour = "black",
) +
scale_y_continuous(
expand = c(0,0),
limits = c(0, (max(Data$FN) + sd(Data$FN))*1.1)
) +
labs(
title = "Graph",
x = "Genotype",
y = "% of positive cells") +
#theme_bw() +
theme_classic() +
theme(
title = element_text(size = 20),
legend.title = element_text(size = 10),
axis.title.x = element_text(size = 15),
axis.title.y = element_text(size = 15),
axis.text.x = element_text(size = 12, colour = 1, angle = 45, hjust = 1),
axis.text.y = element_text(size = 10, colour = 1),
) +
facet_grid(~ Region, scales = "free")




