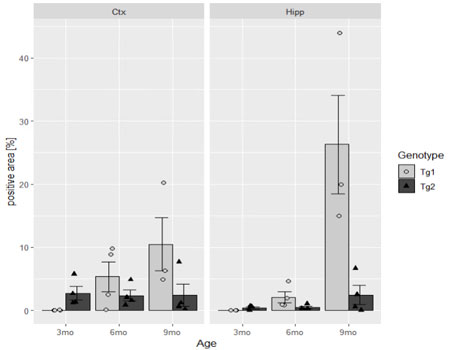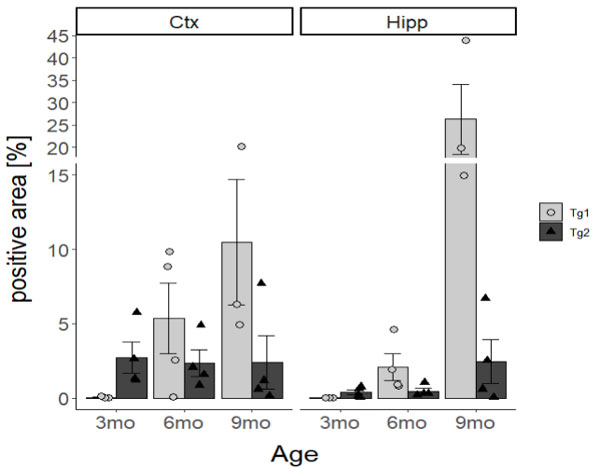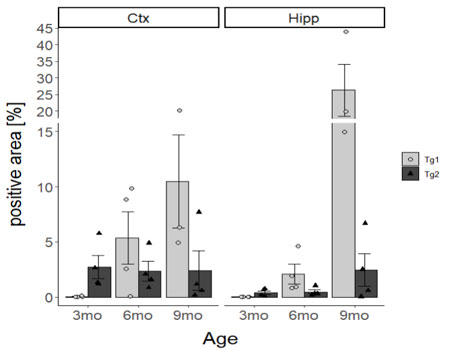
以下に示すのは、ggplotを使って、平均値で棒グラフを作る方法。
前準備
パッケージのインストール
あらかじめ、必要なパッケージをインストールしておく。
library(tidyverse)
library("ggplot2")データフレームのインポート
使用するデータフレームをインポートする。
今回は、任意の場所に作っておいたxlsxファイルを使用。
# xlsxファイルの読み込み
Data1 <- readxl::read_xlsx("D:/R/R directory/Data/XX.xlsx")
# 必要な情報だけ抽出
Data2 <- dplyr::select(Data1,
"Image",
"ID",
"Region",
"Age",
"Genotype",
"positive area [%]")エクセルのデータを R にインポートする方法。 csv ファイル エクセルデータを csv ファイルに保存してインポートするのが最も一般的。 CSV ファイルとは CSV ファイルは、「comma Separated V …
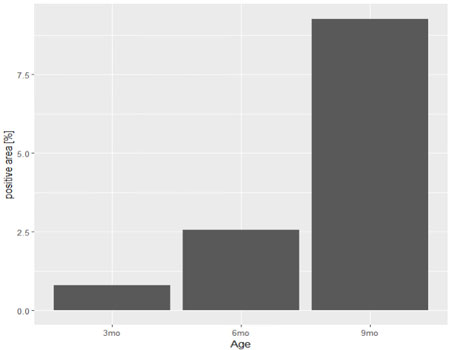
棒グラフを作成
棒グラフは、geom_colでも作れるけど、今回は平均値の棒グラフを作りたいので、stat_summary()を使う。
平均値で棒グラフを作成
# 平均値で棒グラフを作成
ggplot(Data2,
aes(x = Age, # 横軸に"Age"のデータ
y = `positive area [%]`)) + # 縦軸に”positive area [%]のデータ
stat_summary(
fun = "mean", # 平均値で作成
geom = "bar" # 種類は棒グラフ
)
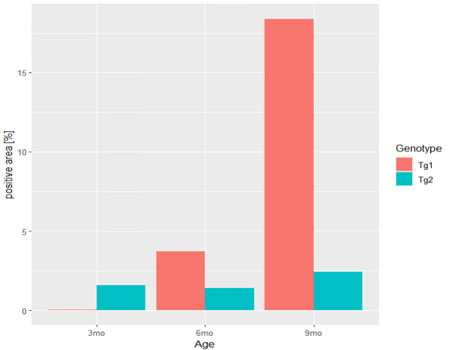
2群で並べる
上のグラフは、2つのGenotype(Tg1とTg2)のデータが合わさって描出されている。
これを、Tg1とTg2のデータで分けて横並びに表示する。
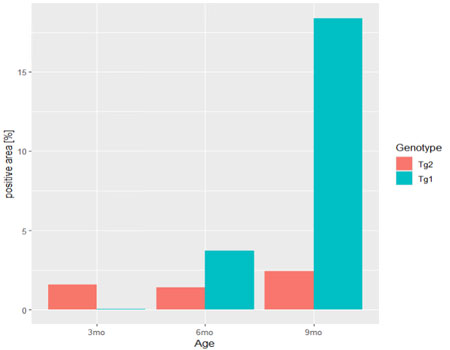
Tg2→Tg1の順序を、Tg1→Tg2と並び替えたいので、データフレームのGenotypeの列の要素をファクターに変え、レベルをつける
# Tg1→Tg2の順に変更
Data2$Genotype <- factor(Data2$Genotype, levels=c("Tg1", "Tg2" ))
# strで確認
str(Data2)無事にGenotypeにレベルをつけられた。
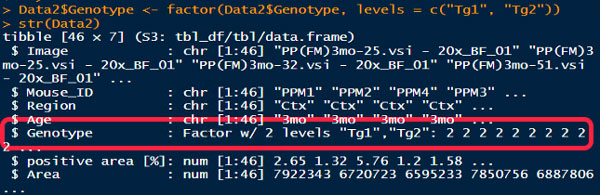
これで先ほどのコードを再度実行すると、Tg1→Tg2の順に入れ替わった。
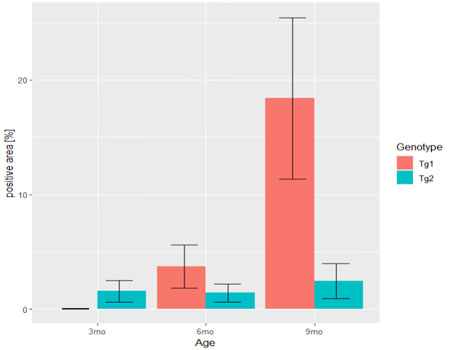
エラーバーをつける
エラーバーをつける。
今回はSEMで。
# errorbarをつける
stat_summary(
fun = "mean", # エラーバーの中心は平均値
fun.min = function(x)mean(x) - sd(x)/sqrt(4), # SEM(上)
fun.max = function(x)mean(x) + sd(x)/sqrt(4), # SEM (下)
geom = "errorbar", # エラーバーを指定
position = position_dodge(.8), # エラーバーの配置
width = .5, size = .1, # エラーバーの太さと幅
colour = "black" # エラーバーの色
)
グラフの棒の色を変更
棒の色をグレースケールに変更する。
ggplot(Data2,
aes(x = Age,
y = `positive area [%]`,
fill = Genotype)
) +
stat_summary(
fun = "mean",
geom = "bar",
position = "dodge",
colour = "black") + # 棒グラフの枠を黒に
scale_fill_manual( # 棒グラフの中の色を変更
values = c("#CCCCCC","#444444"))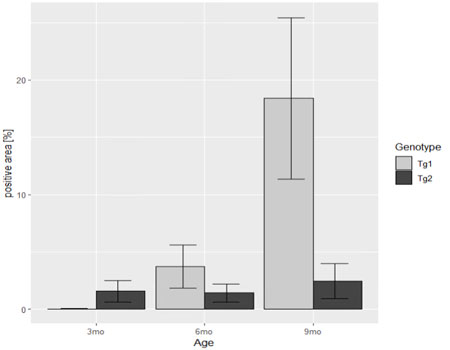
個々のデータをプロットする
平均値とSD(SEM)だけじゃなくて、個々のデータもプロットする。
使うのは、geom_point()
# 個々のデータをプロットする
geom_point(
position = position_jitterdodge(
jitter.width = .3, # プロットの横の散らばり具合を .3 に設定
jitter.height = 0 # プロットの縦の散らばり具合は絶対 0 に。じゃないと嘘のプロットを表示することになる。
),
size = 2, # プロットのサイズ
colour = "black", # プロットの線の色
aes(shape = Genotype) # プロットの形をどのデータでわけるか
) +
scale_shape_manual( # プロットの形をマニュアルで設定
values = c(21, 0)
)プロットの形は、下記参照。
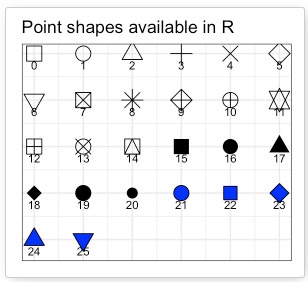
- 0-4 は白抜き。
- 5-20 は黒で塗りつぶし。
- 21-25 はマニュアルで色を設定(色は棒グラフと同じ色)。
今回は、Tg1には21番、Tg2には17番を選択。
色は、先にしてしていた scale_fill_manual() の色で表示される。
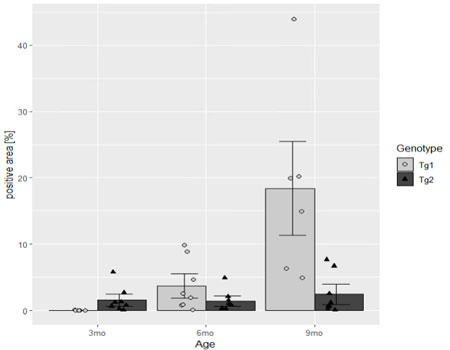
群を分ける
上のグラフは、「Region」の列の「Cox」と「Hipp」のデータが合わさったもの。
これを、「Ctx」と「Hipp」で別々のグラフに表したい。
この時は、Facet()を使う。
# Region毎でグラフを分けて表示
facet_wrap(~Region)CtxとHippで分けて表示された。
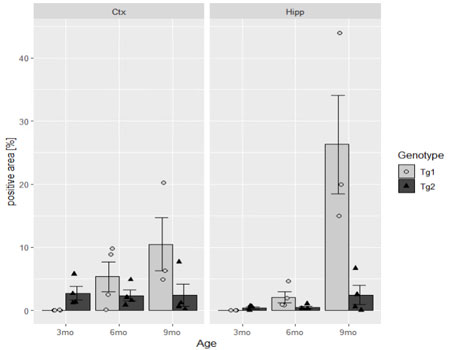
グラフの背景を変更
themeを使って、グラフの背景を変更する。
今回は真っ白にしたかったので、theme_classic()を選択。
# グラフの背景を変更
theme_classic()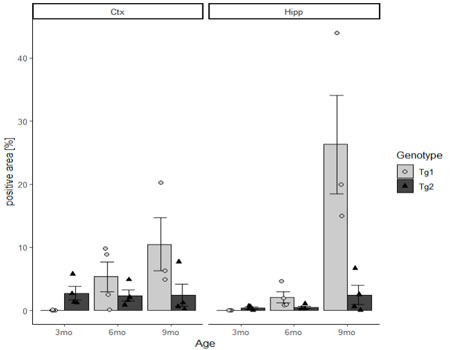
背景が白になった。
他にも色々選択できる。
下のようなグラフがあって、この背景を変更したいとき。 themeで変更する。 グラフの背景の変更 白 theme_classic() グレー+格子(白) theme_gray() 白+格子(グレー) theme_bw() …
Y軸を部分的に切断し、上下で軸幅を変更する
一部の高いバーのせいで、低いバーが見えにくくなっているので、Y軸の「15-19%」あたりで切断し、上の部分の軸幅を半分にする。
# Y軸の「15-19%」あたりで切断し、上の部分の軸幅を半分に
scale_y_break(c(15,19), scale = 0.5)
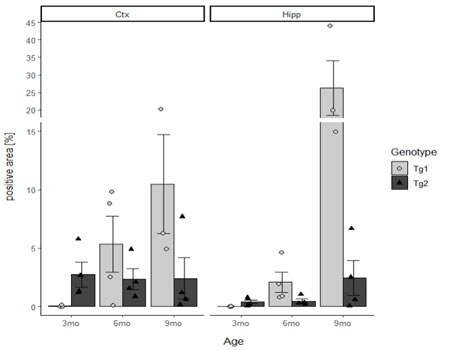
詳細は下記参照。
ggplotで作った下記グラフ、一番高いバーのせいで、低いバーが見えにくくなっている。 なので、途中で棒をぶった切って、中間部を省略し、さらに上の方は間隔を狭めたい。 ……と思った時の備忘録。 軸を切断して途中を省略し、 …
文字の大きさを変更
軸タイトルやレジェンドタイトル等を変更。
# 文字の大きさを変える etc.
theme(axis.title.x = element_text(size = 20, hjust = .45), # X軸のタイトルのフォントサイズと位置を変更
axis.title.y = element_text(size = 20), # Y軸のタイトルのフォントサイズと位置を変更
axis.text.x = element_text(size = 15), # X軸のフォントサイズと位置を変更
axis.text.y = element_text(size = 15), # Y軸のフォントサイズと位置を変更
legend.title = element_blank(), # レジェンドタイトルを消す
strip.text.x = element_text(size = 15) # レジェンドのフォントサイズと位置を変更
)
詳細は下記参照。
下記の様に棒グラフを作成。 # bar graphs Data$Group
コード全体
今回書いたコードの全体像は下記。
# 平均値で棒グラフを作成
ggplot(Data2,
aes(x = Age, # 横軸に"Age"のデータ
y = `positive area [%]`, # 縦軸に”positive area [%]のデータ
fill = Genotype)) + # Genotypeで分ける
stat_summary(
fun = "mean", # 平均値で作成
geom = "bar", # 種類は棒グラフ
position = "dodge", # Genotypeで分けた棒を横並びにする
colour = "black" # 棒グラフの枠を黒に
) +
# 棒グラフの中の色を変更
scale_fill_manual(
values = c("#CCCCCC","#444444") # Tg1とTg2のバーの色を指定
) +
# エラーバーをつける
stat_summary(
fun = "mean", # エラーバーの中心は平均値
fun.min = function(x)mean(x) - sd(x)/sqrt(4), # SEM(上)
fun.max = function(x)mean(x) + sd(x)/sqrt(4), # SEM (下)
geom = "errorbar", # エラーバーを指定
position = position_dodge(.8), # エラーバーの配置
width = .5, size = .1, # エラーバーの太さと幅
colour = "black" # エラーバーの色
) +
# 個々のデータをプロットする
geom_point(
position = position_jitterdodge(
jitter.width = .3, # プロットの横の散らばり具合を .5 に設定
jitter.height = 0 # プロットの縦の散らばり具合は絶対 0 に。じゃないと嘘のプロットを表示することになる。
),
size = 2, # プロットのサイズ
colour = "black", # プロットの線の色
aes(shape = Genotype) # プロットの形をどのデータでわけるか
) +
# プロットの形をマニュアルで設定
scale_shape_manual(
values = c(21, 17)
) +
# Region毎でグラフを分けて表示
facet_wrap(~Region) +
# グラフの背景を変更
theme_classic() +
# Y軸の「15-19%」あたりで切断し、上の部分の軸幅を半分に
scale_y_break(c(15,19), scale = 0.5) +
# 文字の大きさを変える etc.
theme(axis.title.x = element_text(size = 20, hjust = .45), # X軸のタイトルのフォントサイズと位置を変更
axis.title.y = element_text(size = 20), # Y軸のタイトルのフォントサイズと位置を変更
axis.text.x = element_text(size = 15), # X軸のフォントサイズと位置を変更
axis.text.y = element_text(size = 15), # Y軸のフォントサイズと位置を変更
legend.title = element_blank(), # レジェンドタイトルを消す
strip.text.x = element_text(size = 15) # レジェンドのフォントサイズと位置を変更
)