相変わらず R でデータ整理中。
今回は、データベースに記載されている 1+, 2+ などのスコアを数値 1, 2... に変換し、データ型を数値型に変更する方法。
データベースから落としてきたときは、下記のようになっている。
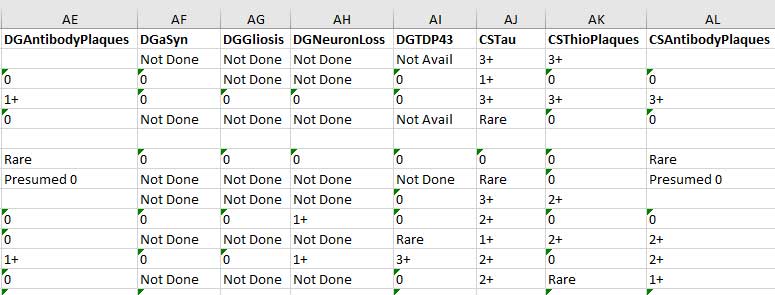
これをそのまま R に取り込むと、文字列として認識される。
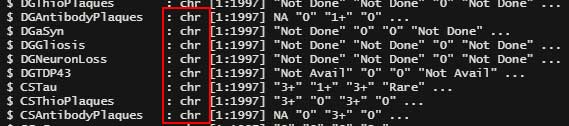
全ての文字列を数値化し、中央値とか出しておきたい。
データフレーム内の特定の文字列全てを数値に変換
まずは文字列を、下記のように変更したい(エクセルの検索→置換機能に該当)。
- Presumed 0 -> "0"
- Rare -> "0.5"
- 1+ -> "1"
- 2+ -> "2"
- 3+ -> "3"
- Not Done -> "NA"
- Not Avail -> "NA"
方法はいくつかあるけど、私は
dplyr::mutate_allstr_replace# Get the data from database
Data <- readxl::read_xlsx("./Demographic_analysis/INQuery_Output-1.xlsx", range="A1:FK1998" ) %>%
# Change the score from character to number
dplyr::mutate_all(~str_replace(., "\\+", "")) %>%
dplyr::mutate_all(~str_replace(., "Rare", "0.5")) %>%
dplyr::mutate_all(~str_replace(., "Presumed 0", "0")) %>%
dplyr::mutate_all(~str_replace(., "Not Done", "NA")) %>%
dplyr::mutate_all(~str_replace(., "Not Avail", "NA"))解説:共通するパターンを変換
1+, 2+, 3+ は、共通する "+" を消したいので、
str_replace(x, pattern = " ", replacement = " ")ここで注意すべきは、"+" は正規表現に使われるので、バックスラッシュ2個 "\\" でエスケープする必要がある。
正規表現については下記参照 ▼
色々使える正規表現の覚え書き。 正規表現(Regular Expression, Regex)とは 正規表現とは、「いくつかの文字列を一つの形式で表現するための表現方法」。 この形式を使えば、違う言葉etc.が入った文字 …
gsub()その際も、正規表現記号にはエスケープが必要(下記も参照 ▼)。
データ整理の続き。 とある csv データを取り込み…… Data1
文字列型を数値型等に変換
上述の操作を行うと、データ型が全て文字列型に変換されるので、必要に応じて数値型や因子型などに変換する。
Data <- Data %>%
mutate(BrainWeight = as.numeric(BrainWeight)) %>%
mutate(Braak03 = as.numeric(Braak03)) %>%
mutate(ABeta = as.numeric(ABeta)) %>%
mutate(Braak06 = as.numeric(Braak06)) %>%
mutate(CERAD = as.numeric(CERAD)) %>%
mutate(Age = as.numeric(Age)) %>%
mutate(GlobalAgeOnset = as.numeric(GlobalAgeOnset)) %>%
mutate(DiseaseDuration = Age - GlobalAgeOnset) %>%
mutate_at(vars(ends_with("Tau")), funs(as.numeric)) %>%
mutate_at(vars(ends_with("ThioPlaques")), funs(as.numeric)) %>%
mutate_at(vars(ends_with("AntibodyPlaques")), funs(as.numeric)) %>%
mutate_at(vars(ends_with("aSyn")), funs(as.numeric)) %>%
mutate_at(vars(ends_with("Gliosis")), funs(as.numeric)) %>%
mutate_at(vars(ends_with("NeuronLoss")), funs(as.numeric)) %>%
mutate_at(vars(ends_with("TDP43")), funs(as.numeric))解説:特定の文字が含まれる列のデータ型を一気に変換する方法
11-17行目までのように、ひとつひとつのデータを
as.○○今回の場合は、各脳部位のスコアを全て取り出していて、
- AmyTau
- DGTau
- CSTau
- ECTau …
のように、
- Tau
- ThioPlaques
- AntibodyPlaeues
- aSyn
- Gliosis
- NeuronLoss
- TDP43
の名前が入った項目が16ヶ所の脳部位の数だけ存在する。
これを一つ一つ変換していったらコードが長くなるので、
end_withData <- Data %>%
mutate_at(vars(ends_with("Tau")), funs(as.numeric)) %>%
mutate_at(vars(ends_with("ThioPlaques")), funs(as.numeric)) %>%
mutate_at(vars(ends_with("AntibodyPlaques")), funs(as.numeric)) %>%
mutate_at(vars(ends_with("aSyn")), funs(as.numeric)) %>%
mutate_at(vars(ends_with("Gliosis")), funs(as.numeric)) %>%
mutate_at(vars(ends_with("NeuronLoss")), funs(as.numeric)) %>%
mutate_at(vars(ends_with("TDP43")), funs(as.numeric))
変換後、
str()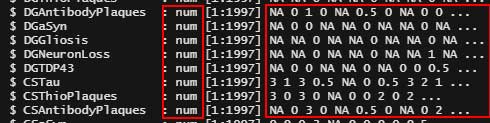
無事に置換 → データ型変換が実行された。
因子型への変換
文字列から因子型への変換は
データ$列 <- factor(データ, levels=c( "レベル1" , "レベル2", ... ))# Change to factor
Data$Sex <- factor(Data$Sex, levels=c(
"Male" ,
"Female"
))数値型として認識されている列は、一度文字列に変換してから因子型に変換する必要がある。
データ$列 <- plyr::revalue(as.character(データ$列), c(レベル1, レベル2, ...)) %>%
factor(levels = c(レベル1, レベル2, ...)))# Change to character then change to factor
Data$HS <- plyr::revalue(as.character(Data$HS), c(
"No"="HS(-)" ,
"Yes"="HS(+)"
)) %>%
factor(levels = c(
"HS(-)" ,
"HS(+)"
))
Data$LBDSubtype <- plyr::revalue(as.character(Data$LBDSubtype), c(
"N/A" = "No Lewy pathology"
)) %>%
factor(levels = c(
"No Lewy pathology" ,
"Brainstem Predominant" ,
"Amygdala Predominant" ,
"Transitional or Limbic" ,
"Diffuse or Neocortical"
))
Data$LATE <- plyr::revalue(as.character(Data$LATE), c(
"No"="LATE(-)" ,
"Yes"="LATE(+)"
)) %>%
factor(levels = c(
"LATE(-)" ,
"LATE(+)"
))下記も参照 ▼
R にデータを取り込んで、そこから最初に行う作業の備忘録。 データのインポート 私は、データベースからエクセル (.xlsx) でデータを取得し、それを R に取り込む場合が多い。 エクセルの取り込みは、 readxl: …
データ型とデータ構造の覚え書き。 データ型 データ構造を調べたり変換したりする場合は、下記関数が用意されている。 データ型には以下の関数が用意されている。 データ型 データ型を調べる データ型を変換する モード mode …