脳内にある、特定の遺伝子やタンパクの発現量をみるとき、いくつか方法があるけど、個人的によく使うサイトを書き留めておく。
主にはmRNAレベル。
脳の部位別発現量をみたいとき
使っているのは「Allen Brain Atlas」
Accelerating progress toward understanding the brain
マウスの場合
マウスのページを開く
ページの検索欄に、確認したい遺伝子名を入れて「Search」をクリック。
coronal, sagital など、色々な in situ の結果が確認できるので、目的のものを一つ選択。
右上のパネルで、実際の in Situ の画像が確認でき、その下のグラフで、部位別の発現量の比較を確認できる。
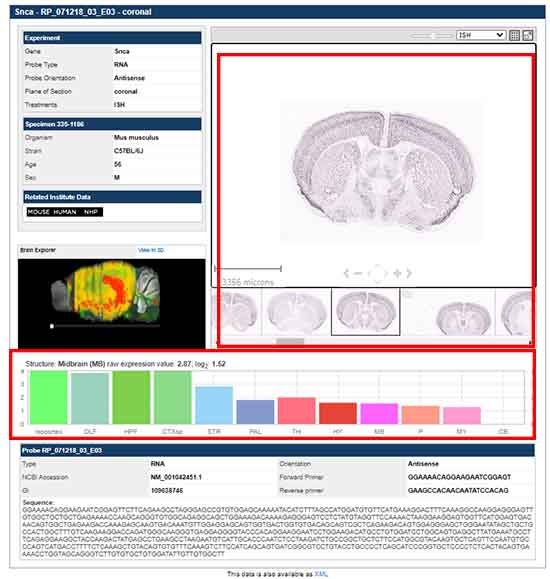
上の例は、SNCA の発現量をみている。等皮質、嗅球、海馬、新皮質、線条体、淡蒼球、線条体、視床下部、中脳、橋、延髄……etc. に発現が多く、小脳にはほとんど発現がないのがわかる。
ヒトの場合
上のタブを開き、「HUMAN BRAIN」を選択。
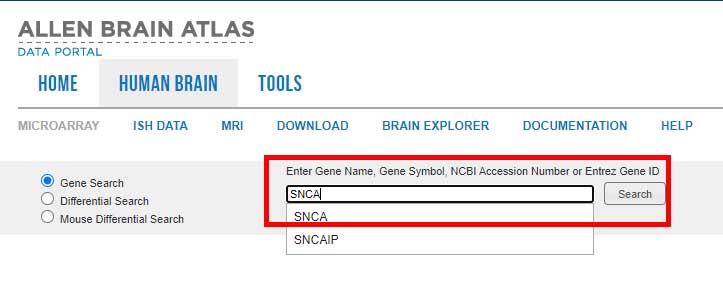
検索欄に確認したい遺伝子名を入れて「Search」をクリック。
下にある「Color Map」で色を選べる(個人的には 青 → 赤 が一番見やすいと思う。)
右のパネルに、何人かのドナーのデータが現れ、それぞれの脳部位別の発現量がカラー表示される。
下記の場合は、ドナーは6人いて、それぞれ皮質や海馬で発現が多い(赤が多い)、というイメージ。
それぞれの場所にカーソルをあわせると、ドナー番号や脳部位が上に表示される。
(下記の例は、H0351.2002のドナーの帯状回を見たときのデータ、という事。)
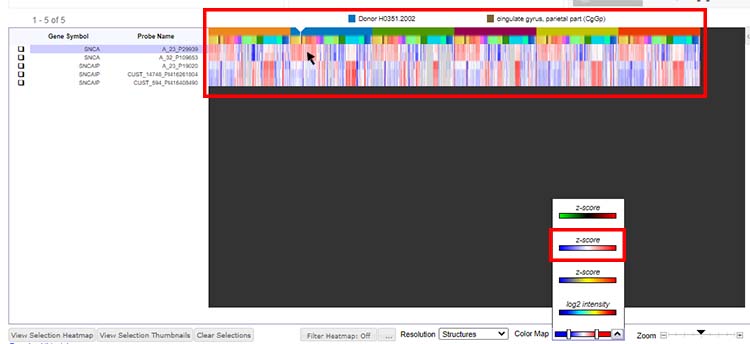
右下のバーで、カラーマップを拡大・縮小しながら確認する。
脳細胞別の発現量を見たいとき
「Brain RNA-Seq」 が使いやすいと思う。
左上の検索画面に確認したい遺伝子名を入れて暫く待つと、候補遺伝子が下にでてくるので、そこから選択する。
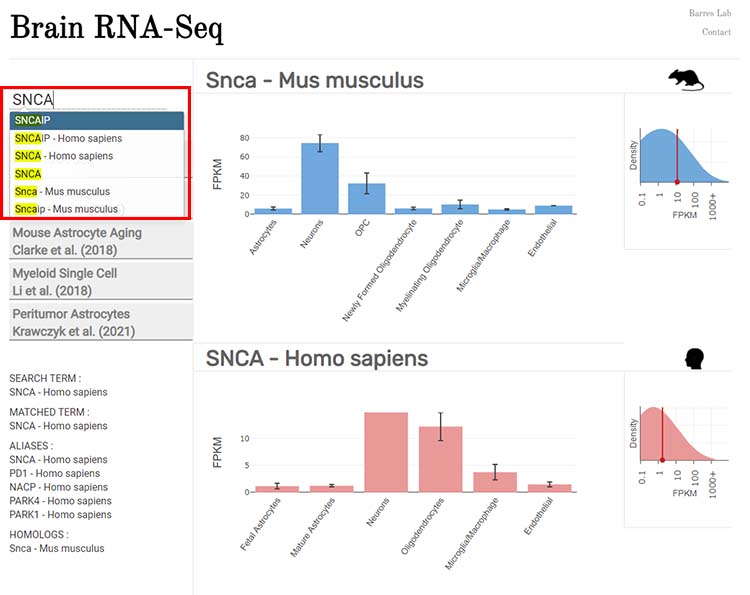
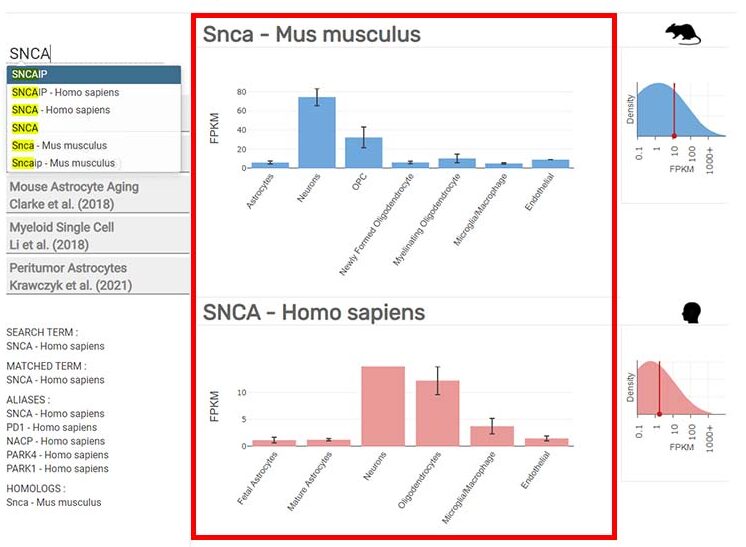
上にマウスのデータ(青)、下にヒトのデータ(ピンク)が表示される。
(下記の例だと、SNCAは、マウスではニューロン > OPC の順に多くて、ヒトだと ニューロン > オリゴ > ミクログリア、マクロファージ の順に多い、という事。)
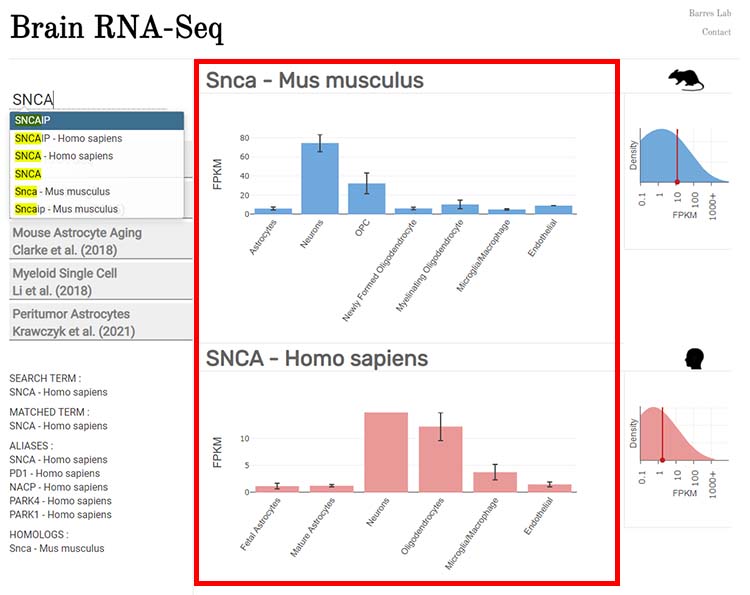
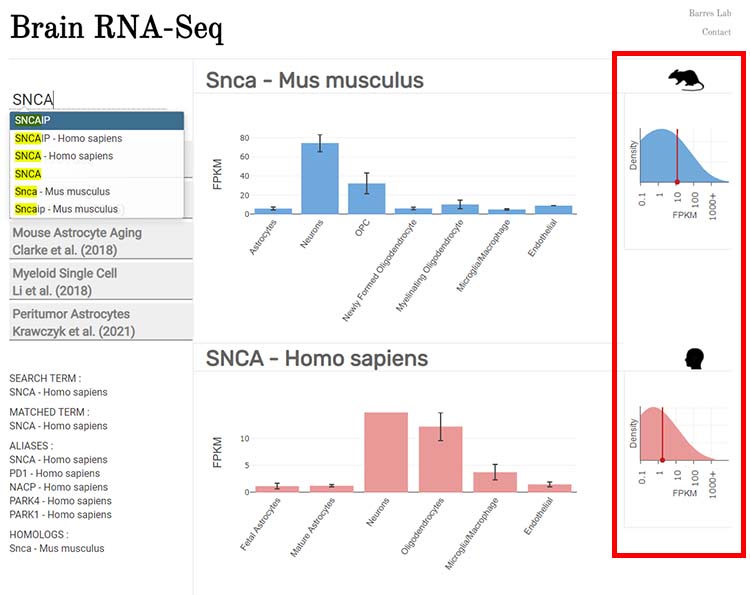
Y軸の FPKM は絶対値を示している。
更に右側にある分布と数値で、絶対的な発現量をみることができる。
(下記の例だと、マウスでは SNCA の発現はまあまあ多いけど、ヒトでは結構少ない事がわかる。)
細胞内小器官別の発現量を見たいとき
使っているのは「Uniprot」
The mission of UniProt is to provide the scientific community with a comprehensive, high-quality and freely accessible resource of protein sequence and functional information.
検索画面に目的の遺伝子名を入れてクリック。

例えば「SNCA」と入れると、Homo sapiens (Human), Mus musclus (Mouse), Bos taurus (Bovine)... と、色々な動物種が出てくるので、目的の種を選択(ここでは Human とした)。
個別画面には、その分子の機能や分子量、結合サイト、変異やその機能、PTM、相互作用分子...と、色々な情報が出てくる。
細胞内の分布は、真ん中あたりにある。
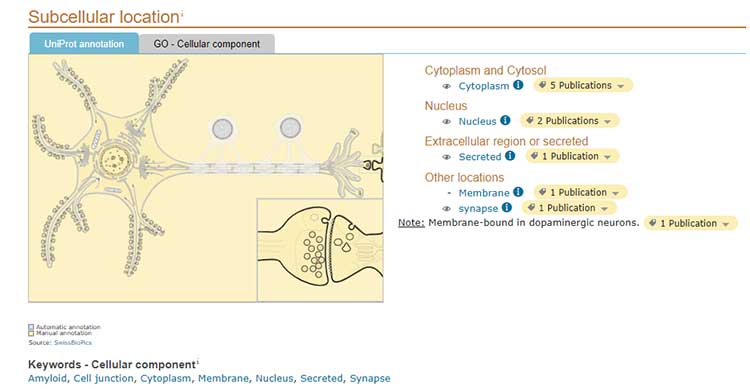
↑ の情報によると、SNCA (α-Synuclein) は、細胞外、核、シナプス、細胞膜に存在している事がわかる。
"Publications" をみると、References を確認することができる。
おまけ:Allen Brain Atlas で、脳の解剖を確認
Allen Brain Atlas は、遺伝子発現以外に、通常の解剖を確認するときによく使っている。
「REFERENCE ATLAS」を開くと、
- 生体マウス
- 成人
- マウス胎仔
- ヒト胎児
- マウス脊髄
- 霊長類
などの解剖を確認することができる。