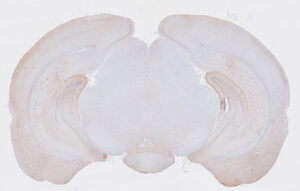
例えば、このマウスのIHC画像。
海馬と皮質の部分をそれぞれannotationして、二値化してDAB染色陽性部分の面積を測定したい。
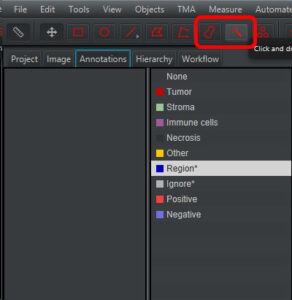
アノテーションを作成する
まずは、計測したいエリアを選択して、アノテーションを作成する。
左タブから「Annotations」タブを選択。
アノテーション作成によく使うのは、赤枠で囲った「Brush」ツールと「Wand」ツール。
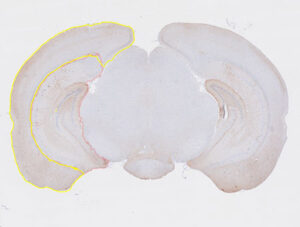
まずはWandツールを使って、輝度値を認識してエリア選択。
でも、完全にはできないから、部分的にBrushツールを使って綺麗に補正。
「Shift」と押しながら左クリックするとinclusion、「Alt」を押しながら左クリックするとexclusionになる。
……こんな感じでアノテーションが作成できた。
二値化して閾値以上の部分の面積を測る
エリアをアノテーションできたら、「Classify」→「Pixel classification」→「Create thresholder」へ進む。
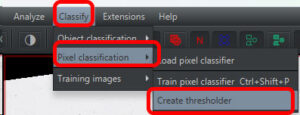
「Create thresholder」のウィンドウが開く。
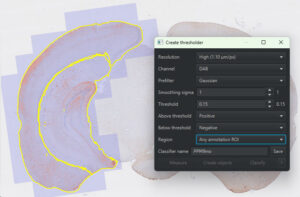
上のバーで透明度を変化させながら、良い具合にThresholdを設定する。
「Create thresholder」パネルの詳細設定
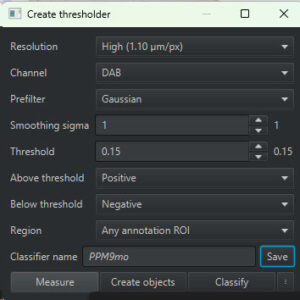
- Resolusion:解像度。Lowにすれば粗く、Highにすれば細かく拾える。データが重くなるのでできるだけ低くしたいが、今回はHigh(1.10um/px)でちょうどバックグラウンドと陽性細胞を分けることができた。
- Channel:今回はDABで。蛍光染色のときは、それぞれのチャネル(Red, Green, Blue)を選択。
- Prefilter:Gaussianが標準。
- Smoothing sigma:シグマ値をスムージングしてノイズを抑える。今回は1くらいがちょうどよかった。
- Threshold:Thresholdを調整する。DABの場合、RBG値の範囲(0-255)とは異なる。今回は0.15くらいがちょうどよかった。
- Above threshold:Positive(陽性所見)
- Below threshold:Negative(陰性所見)
- Region:アノテーションをいくつか作った場合は「Any annotation ROI」にすると一変に計測してくれる。
- Classifier name:適当に名前をつける。
最後に「Save」を押して、Classifierを保存する場所を決めて保存する。
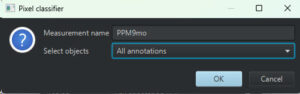
陽性部分を計測
Classifierを保存したら、「Measure」を押して計測。
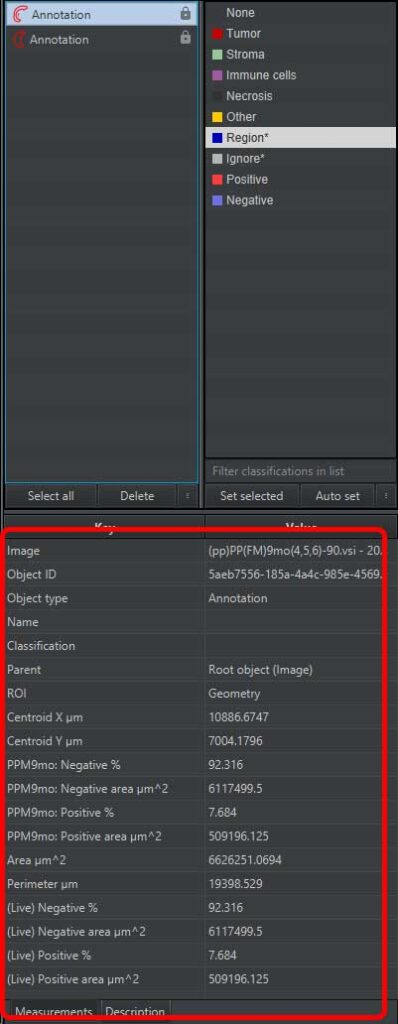
左下に計測結果が表示される。
結果を保存
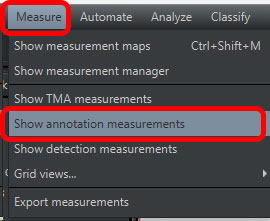
「Measure」→「Show annotation measurements」を選択。
結果のリストが現れる。
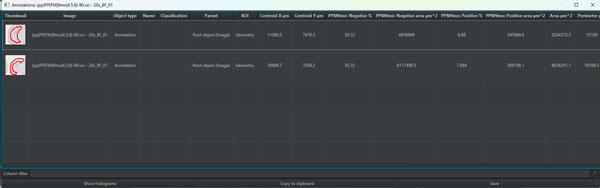
「Save」を押すと、テキストファイルで保存される。